最終更新:2022年 11月 29日 (火曜日)
fmsb-0.7.4(2022年11月29日)
公衆衛生学講義の老人保健の資料を更新するために必要だったので、Jlifeデータフレームにqx2020M及びqx2020Fとして第23回完全生命表(2020年)の変数を追加し、ついでにJfertデータフレームにASFR2020及びASMFR2020として2020年の15-54歳女性の年齢別出生率と年齢別有配偶出生率の変数を追加してfmsbパッケージを更新した。RStudioのcheck Buildではエラーがでなかったので、Build SourceとBuild Binaryを実行し、fmsb-0.7.4.tar.gzをcranにsubmitした。
すると2日ほどで返事があり、別の関数のhelpでの参照先が2つデッドリンクになっているから直して再度投稿せよと書かれていた。かつてデータソースをダウンロードしたURLがデッドリンクになることは、これまでの更新でも度々あったが、今回はインドのセンサスサイトの構造変更だったからリンク変更で済んだ(もう1件は必須ではない参考ページをReferecensに入れていただけだったので削除した)。
しかしこれ、サイトの構造変更ではなく、完全消滅した場合はどうするんだろう? と少し気になったが。
再投稿したら無事にfmsb-0.7.4がCRANに入ったようだ。もっとも、今回はデータ追加だけなので、それらを使わない人はとくにアップデートする必要はない。
R-4.2.2リリース(2022年10月31日)
tweetしたのだけれども、R-4.2.2がリリースされた。コード名であるInnocent and Trustingの出典は、例によってPeanutsの1962年10月30日の回。
Epi R Handbook日本語版(2022年10月15日)
本日 Epi R Handbook の日本語版「疫学のための R ハンドブック」が公開されました!! 読んだ感想や使用例を自由に発信してくださいとtweetされた通り、日本語版「疫学のための R ハンドブック」が読める。ここに書かれている方法で、オフライン版も利用できるようだ。目次や推奨パッケージを見ると、感染症疫学や空間疫学や遺伝疫学もカバーしているようだが、tidyverseとgridグラフィクスなのが個人的には残念。かなり丁寧に説明されている人口ピラミッドはapyramidパッケージよりも拙作pyramidパッケージで作る方がきれいだと思う(個人の感想……というか、審美眼は主観なので、もちろん異論は認めるが、ぼくはbaseグラフィクスの方が好きだし操作しやすいと思うので)。
DescToolsパッケージは便利(2022年10月15日)
長崎大学の高橋先生のこのtweetで、normtestパッケージがcranからインストールできなくなっていることを知った(注:以前触れたLillieforsの検定が入っているnortestパッケージとは別物)。8年ほど更新がなかったが、今年の6月にcranのチェックを通らなくなってライブラリから外れたようだ。尤も、Archiveにはバージョン1.1のソースが残っているから、たぶん頑張ればそこからインストールできると思う。
しかし、R界隈では頑張らなくても別解がある場合が多く、normtestパッケージに入っていたジャック・ベラの正規性検定をするには、DescToolsパッケージに実装されている関数を使うのが楽だ。高橋先生の本と同じ結果を得るには、library(normtest)をlibrary(DescTools)に変え、jb.norm.test(resid1)をJarqueBeraTest(resid1, robust=FALSE, method="mc", N=1000)に変えれば良い。ちなみにDescToolsのJarqueBeraTest()関数は、デフォルトではロバストなジャック・ベラの検定をするので結果が異なるし(Lillieforsの検定に触れたときに参照したUyanto SSの論文―いつの間にかpublishされていたを読むと、ロバストな検定の方が良い気がする)、モンテカルロシミュレーションではなくカイ二乗近似で結果を得る、method="chisq"というオプションもある。
DescToolsは大勢の研究者がgithubで共同開発している大変高機能な記述統計用のパッケージで、DescTools Companionの下書き(2018)を眺めるだけでも、できることが多くて驚くのではないかと思う。R使いならインストールしておくべきだろう。
R-4.2.1リリース(2022年6月26日)
R-4.2.1(コード名はFunny-Looking Kid……直訳すると面白い顔をした子どもという意味だが、Peanutsの出典回と思われる話から考えるとスヌーピーのことらしい)が6月23日付けでリリースされていた。R-4.2.0のWindows版で一時的におかしくなっていたclipboardデバイスへの書き込みがバグフィックスされたとのこと。リンク先の説明を読む限り、RがWindows版も4.2.0からUTF-8ベースで動くようにしたことが、それまで暗黙の仮定で動作していたインターフェースの不具合を浮かび上がらせたという感じか。
R-4.1.3リリース(2022年3月10日)
R-4.1.3がリリースされた。コード名は"One Push-Up"であり、これまで通りPeanutsが出典だとするとこれか? 野球チームのマネージャーになったらしいチャーリー・ブラウンが、シーズン開幕に当たって、まず激しい美容体操筋トレ(注:器具を使わない体操を意味する)から始めようと言ったらたくさんのグラブや帽子を投げつけられたので、「腕立て伏せ1回」はどう? と尋ねるというオチ。R-4.1.3はR-4.1.2に比べて腕立て伏せ1回くらいの微修正ということだろうか。
Lillieforsの検定とコルモゴロフ=スミルノフ検定(2022年3月10日)
COVID-19感染が脳に器質的ダメージを与えるというNature論文に、Lillieforsの検定で有意だったので2群の比較はノンパラでやったと書かれていたので、Lillieforsの検定を検索してみたら、Rではnortestパッケージにlillie.test()として実装されていて、コルモゴロフ=スミルノフ検定(それ自体はR本体にks.test()として実装されていて、例えばxという数値ベクトルオブジェクトが標準正規分布と差が無いという帰無仮説の検定は、ks.test(x, "pnorm")でできる。xが平均0分散1でないときは標準化すれば良い)の改良版らしい。一般論としては、正規性の検定で有意だからノンパラというロジックはお薦めできないが、どちらかといえば保守的になるはずなので、差があったという結果は正しいのだろう。
先日メモした(下記引用)、50種類の正規性の検定を比較した論文を読むと、Lillieforsの検定とコルモゴロフ=スミルノフ検定の違いがわかりやすかった。ちなみに、Lillieforsの元論文を見るとD統計量の計算方法(サイズNの標本データXに対して、その累積分布関数SN(X)と、平均がXの平均値、分散がXの不偏分散である正規分布F*(X)の差の絶対値の最大値をDとする)と1000回のシミュレーションによって求めたp値の表が載っているのだが、nortestパッケージのlillie.test()のD統計量の計算の実装は見かけ上元論文と違っていて(欠損値を除去、小さい順に並べ替え、標準化した値の標準正規分布の分布関数値を計算、1:n/nとの差の最大値と0:(n-1)/nとの差の最大値の大きい方をDとする、となっていて、50種類の正規性の検定を比較した論文でもそちらの説明が与えられているので、たぶんThode Jr. 2002の本でのLillieforsの検定の説明がそうなっているのだろう。得られる結果は同じだが)、p値も表でもシミュレーションでもなく近似多項式で与えるようになっていて、適当な乱数を作って試してみたら、lillie.test()でもks.test()でもD統計量は同じだが、p値がかなり違っていて、ほとんどの場合にlillie.test()のp値の方が遥かに小さくなった。長さ100の正規乱数を100回作って試したら、ks.test()ではp値が0.05未満には一度もならなかったが、lillie.test()では5回前後0.05未満になった。なるほどそういうことか。
Publish前のpreviewだが、Uyanto, S. S. An Extensive Comparisons of 50 Univariate Goodness-of-fit Tests for Normality. Austrian Journal of Statistics, 51(3), 45–97. Retrieved from https://ajs.or.at/index.php/ajs/article/view/1279は面白い。正規性の検定50種類を比較している。fmsbにgeary.test()として実装したGearyの検定も入っている。fBasicsなど、使ったRのパッケージがたくさん挙げられているが(fmsbは入っていなかったので、Gearyの検定は、normtestパッケージのgeary.norm.test()を使っているのだと思われる)、自由度3のt分布、ラプラス分布、locationが0でscaleが4のロジスティック分布、locationが0でscaleが3のグンベル分布、locationが0、scaleが1、slantが7の歪正規分布、shapeが2でscaleが1のガンマ分布、shapeが1.5でscaleが1のワイブル分布、shapeが2と5のベータ分布、0から1までの一様分布(これらの分布関数のいくつかはExtDistパッケージに入っている。歪正規分布はsnパッケージに入っている)に従う乱数について、サンプルサイズを10から100まで5段階で変えて、それぞれ10000回の正規性の検定をして検出力を調べたところ、コルモゴロフ=スミルノフ検定とGearyの検定は有名だけれども検出力は最低の部類で、多くの先行研究ともそれは一致していた、と結論している。表を見ると、標準的に知られている正規性の検定の中では、Shapiro-Wilkの検定がかなり検出力が高い。
かつてはSASとユーザー会のライブラリしかサポートしていない解析手法もあったが、今では最先端の解析の多くはRかPython(Cなどを含むこともあるが)のソースコードかパッケージともに発表されるので、自分で実装する必要はなくなってきた。Gearyの検定も、自分で作ってfmsbに入れたときは、まだサポートしているパッケージがなかったはずだが、隔世の感がある。
fmsb-0.7.3
人口動態統計の2020年確定数が出た。fmsbパッケージ内のJvitalデータを更新しなくてはならないので、ついでにRose Chartをフルスクラッチで書いてみるか? それより大事なことは、0.7.2で入れ損なった、spearman.ci.sas()の欠損値対応を忘れないようにしないと。もっとも、締め切りを延長して貰った採点と成績入力を完了するのが先決だし、3月上旬に第23回完全生命表が発表されるはずなので、Jlifeも同時に更新したいところだが。2020年だとあまり顕著でないが、2021年9月の月報速報を見ると、2021年の死亡増加はかなり顕著なので(2020年9月の月報速報を見ると、2019年と2020年の月別死亡数はほぼ変わっていない)、たぶん最近5年くらいの死因別死亡のレーダーチャートを描くと2021年のCOVID-19の影響がはっきり見えると思う。平均寿命がはっきり短縮した欧米ほどではないにしても、旧専門家会議を解体して分科会を発足させて以降、日本もCOVID-19対策には失敗している。(2022.2.25)
予定したデータ追加と欠損値対応(spearman.ci.sas()だけでなく、geary.test()とtruemedian()にも必要だった)を済ませ、fmsb-0.7.3をビルドしてcranに投稿したら、20:40を過ぎてしまった。(2022.3.1)
fmsb-0.7.3は無事に審査を通ったようで、日付が変わってすぐくらいのタイミングで、以下のメールが届いていたので、たぶん明日くらいまでにはinstall.packages("fmsb")でインストールでき、インストール済みの方はupdate.packages(checkBuilt=TRUE, ask=FALSE)でアップデートできるようになるはず。(2022.3.2)
Dear maintainer,
thanks, package fmsb_0.7.3.tar.gz is on its way to CRAN.
Best regards,
CRAN teams' auto-check service
『データ視覚化の人類史』
『データ視覚化の人類史』(青土社)を偶然見かけ、面白そうだったのでKindle版を衝動買いしてしまった。口絵として有名なSnowのボロノイ図(かつてtweetしたが、Rではcholeraパッケージを使うと簡単に描ける)とかナイチンゲールのrose chart(RではHistDataパッケージに入っているNightingaleというデータセットを使えば描ける。使い方はexample(Nightingale)でわかる。ただしパッケージ名はhistDataではなくHistDataが正しいので注意)が載っていた。HistDataパッケージにはもちろんSnowのデータも入っていて、library(HistData); SnowMap(polygons=TRUE)でボロノイ図が描ける(choleraパッケージで描ける図と見比べると面白い)。当時のロンドンのコレラ死者について標高と給水系統とともにFarrが記録させたデータもCholeraとして入っていてexample(Cholera)とすると解析例が出てくる。疫学教育に大変役に立ちそうなパッケージ。
昨日メモした『データ視覚化の人類史』は、原著がMichael Friendly & Howard Wainer "A History of Data Visualization and Graphic Communication"(Harvard Univ. Press, 8 June 2021)で、Kindle版でも6,000円以上するのだが、それから半年も経たずに邦訳が出て、しかも原著の半額以下というのは、お買い得感が凄い。HistDataパッケージのリファレンスマニュアルを見ると、筆頭開発者にしてメインテナが本書の筆頭著者であるMichael Friendlyさんであることがわかる。マニュアルにはReferencesの中にFriendly, M. & Wainer, H. (in progress). The Origin of Graphical Species. Harvard University Press.と書かれており、これが改題されて出版されたのだと思う。つまり、まったく偶然に、ぼくは本書のサポートパッケージに辿り着いていたわけだ。通勤途中で読み始めたが、著者二人はTukeyの弟子筋だそうで、FriendlyさんのtwitterにはTukeyのお墓の写真が載っていた。
裏RjpWikiさんが、ナイチンゲールの鶏頭図--再びという記事で、HistDataパッケージは「melt がないとか,reshape がないとか,ggplot2 がないとかうるさかった割に,ちょっと残念な出来栄え」として、Juliaで描画した図を掲載している。たぶんRでもgridグラフィクスを使わない方が簡単に書けそうだが、このデータならむしろrose chartよりも、cumsumを使って3重のレーダーチャートにした方が見やすいのではないかと思う。ただしHistDataパッケージはあくまでナイチンゲールが作った図の再現を目指しているので、レーダーチャートを描いては主旨が変わってしまうわけだが。
Nightingaleのrose chartは、baseグラフィクスならばplotrixパッケージのradial.pie()関数を使えば描けるが、たぶん実装上のバグに近いと思うのだけれども、radial.limを指定できるのに描画座標系をradial.extentsで決めているために2つのグラフを同じスケールで描くことができない(あと、labels=オプションがstart=とclockwise=に追従してくれない)。というわけでスケールが変わってしまうが、HistDataパッケージとplotrixパッケージをインストールしてあれば、このコードを実行すると下図が描ける(修正追記:plotrixパッケージのradial.pie()関数は与えた数字が半径として使われるので、裏RjpWikiさんが言われるように面積にしなくてはいけないとすると、工夫が必要だった。ここまでして、かつ、数値ラベルもきれいに打てないなら、フルスクラッチで書いた方が早いかもしれない)。
rosettaパッケージ
井出草平さんの順序α (Ordinal Alpha)[R] で、userfriendlyscienceがufsに変わっていたことを知った。もっと困ったのはposthocTGH()がufsには入っていなかったこと。検索してrosettaというパッケージに入っていることがわかった。弘前大学の改変RコマンダーはGames-Howell法をサポートしているので、とっくにこのことには気づいていて依存パッケージの変更もされていた。R-4.0.5以降の変更とのこと。保健学研究共通特講IV/VIIIのテキストとして作っている「保健・医療研究の進め方入門」の記載も直して1.0.4版とした。
useR!2022
2022年6月20-23日にオンライン開催されるuseR! 2022の事務局からメールで案内が来た。チュートリアル報告申込の締め切りは2月15日、一般報告申込締め切りは3月1日。参加受付は3月1日開始とのこと。公式twitterアカウントをフォローした。
「卒業論文のためのR入門」
立命館大学の森さんがこのtweetで紹介している,「卒業論文のためのR入門」。RとRStudioをインストールしてデータを読み込んで前処理をして記述統計とグラフを作ってt検定と回帰分析をして結果をWordに貼り付けるという順番で説明され,随所に解説動画へのリンクがあって親切だと思う。結果の表の貼り付けに関しては,LaTeX形式やhtml形式で出力するパッケージはtablesとかstargazerとかsummarytoolsとかxtableとかたくさんあるし,EZRの結果のサマリー表の出力メニューを実行したときにクリップボードにコピーされた情報をExcelに貼り付けると表への加工はやりやすいのだが,Wordに直接貼り付けるパッケージは知らないなあ(いったんコンマ区切りかタブ区切りテキストとしてペーストして表にコンバートという方法を説明しているページが多いが,Wordを持っていればExcelも持っているだろうから,Excelに貼り付けて横罫線を入れてからコピーしてWordにペーストする方が楽だと思う)。検索してみたら,ReporteRsというパッケージがあったが。この点,jamoviだと解析結果をコピーしてWordにペーストするだけでちゃんと表になっているから,卒論生にはjamoviの方がずっとハードル低いだろうなあ。保健学研究共通特講IV/VIIIのテキストのサブテキストとして,R/EZRで説明してある解析をjamoviでやるには? という文書を作るべきかもしれない(ちょっと作り始めたら,追加モジュールとしてRjエディタを呼び出してRコードを実行する際に,System Rを呼び出すこともjamovi Rでパッケージをインストールすることもうまく行かなかったので,fmsbパッケージに入れてあるriskratio()を実行させるには素のRでlibrary(fmsb); riskratioと打てば表示されるriskratio()のソースコードをRjエディタ画面にコピー&ペーストするしかなかった……これはWindows版だけの問題なのだろうか?)。
Draperyプロット(p値関数)
p値関数はmetaパッケージのdrapery()関数のtype="pval"で描けるんだな。メタアナリシスの各研究のp値関数と要約統計のp値関数を重ね描きしてくれるようだ。疫学特講の第10回で10研究からのメタアナリシスと11研究からのメタアナリシスのp値関数を重ね描きするため,pvpORMH()という関数を作ってfmsbにも入れたが,メタアナリシスではフォレストプロットの追加としてDraperyプロットも描いた方が良いとされているようだ。
1月2日に触れたmetaパッケージの使い方をメモしておく。Rを起動して,まず,以下を打ってメタアナリシスのサンプルデータTenStudiesとElevenStudies(内容は疫学講義第10回のスライド9を参照)をロードする。
library(meta)
library(fmsb)
example(pvpORMH)
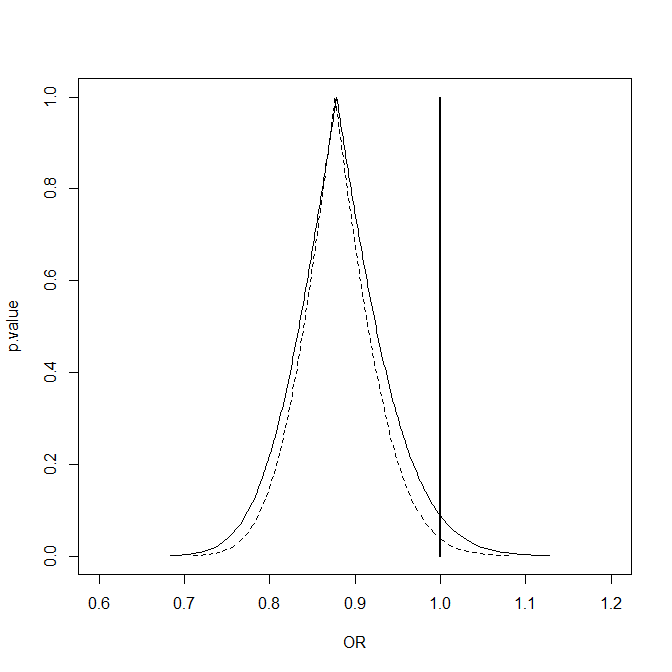
Rothman KJ et al. (1999) Lancetに載っているのと同じ,上のグラフが描かれる。ここで10個の研究についてのメタアナリシスとして,マンテル=ヘンツェルの要約オッズ比を計算するには,fmsbでは,ORMH(TenStudies)で良い。出力もシンプルで,
$estimate
[1] 0.8781804
$conf.int
[1] 0.756586 1.019317
$conf.level
[1] 0.95
となる。metaでは,
res <- metabin(TenStudies[,1],TenStudies[,1]+TenStudies[,3],TenStudies[,2],TenStudies[,2]+TenStudies[,4], sm="OR")
res
とする。fmsbパッケージのORMH()は要約統計量としてオッズ比しか計算できないが,metaパッケージのmetabin()は,sm="RR"と指定すればリスク比を,sm="RD"とすればリスク差を計算できる(もちろん元データがコホート研究かRCTでなくては無意味だが)。
プール化の方法も,ORMH()はマンテル=ヘンツェルの方法だけだが,metabin()ではmethod="Inverse"オプションにより逆分散重み付け法(Fleiss, 1993),method="Peto"でPetoの方法(Yussuf et al., 1985),method="SSW"でサンプルサイズ法(Bakbergenuly et al., 2020)が適用される。たぶん本来はmatrix()ではなく,data.frame()に対して使うように作られていて,data=オプションでデータフレーム名を指定すれば,引数としては,曝露群イベント発生数,曝露群サンプルサイズ,非曝露群イベント発生数,非曝露群サンプルサイズの順番に変数名を書けば良い。studlab=オプションで研究名のラベルを示す変数名を与えることもできる。結果は以下のように得られる。Huedo-Medina T et al. (2006)によると,コクランのQ統計量を使ったQ検定でもI2指標を使っても研究数が少ない場合の欠点である異質性の検出力の小ささは解決できないとのことだが,この程度の数値ならば,有意な異質性はなく,マンテル=ヘンツェルの方法で固定効果モデルでプール化した統計量としての要約オッズ比を出しても良いと考えられる。
Number of studies combined: k = 10
Number of observations: o = 3088
Number of events: e = 1771
OR 95%-CI z p-value Common effect model 0.8782 [0.7566; 1.0193] -1.71 0.0876 Random effects model 0.8782 [0.7563; 1.0198] -1.70 0.0885 Quantifying heterogeneity:
tau^2 = 0 [0.0000; 0.0948]; tau = 0 [0.0000; 0.3078]
I^2 = 0.0% [0.0%; 62.4%]; H = 1.00 [1.00; 1.63]
Test of heterogeneity:
Q d.f. p-value
6.95 9 0.6422
Details on meta-analytical method:
- Mantel-Haenszel method
- Restricted maximum-likelihood estimator for tau^2
- Q-profile method for confidence interval of tau^2 and tau
ORMH()の結果とCommon effect modelの結果が一致していることがわかる(マンテル=ヘンツェルの方法とPetoの方法は固定効果モデルを前提としているが,変量効果モデルでの計算結果も出力される)。
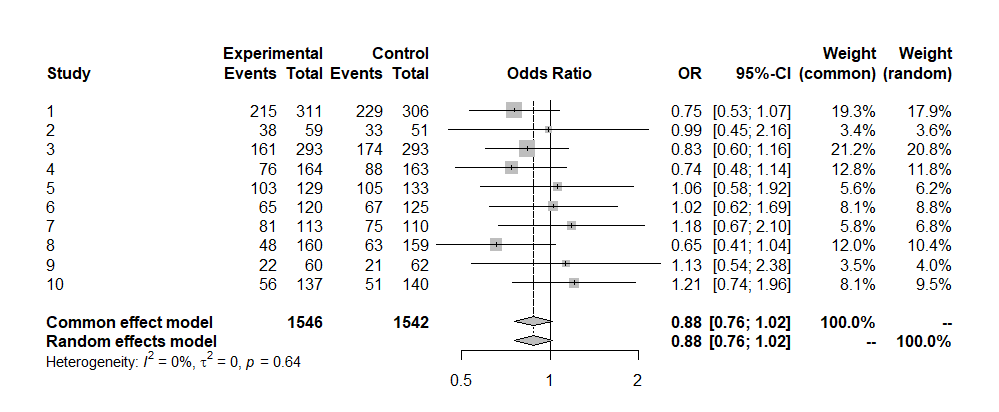
metaパッケージが偉いのはここからで,forest(res)とすると以下のForest Plotが描かれる。
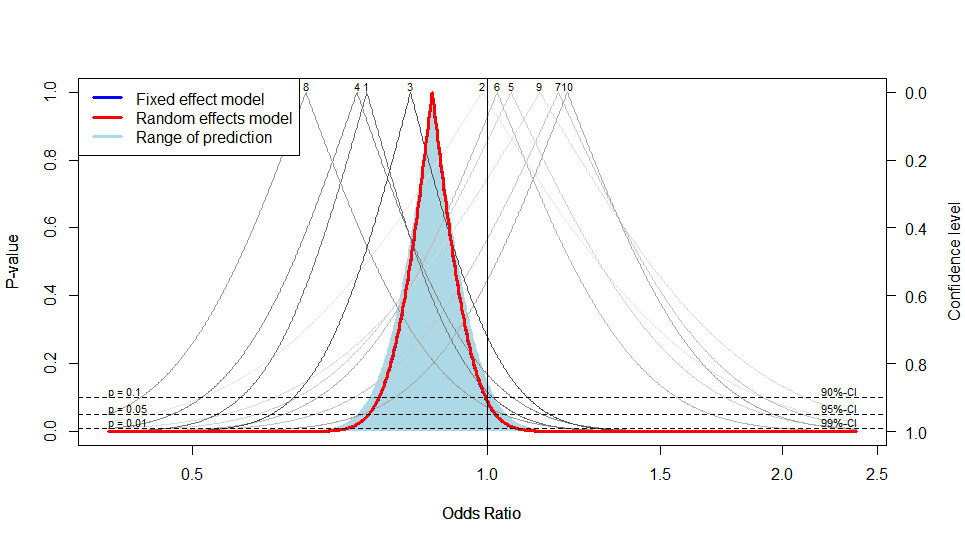
さらに,drapery(res, type="pval")とすると,10個の研究それぞれについてのp値関数がグレーで,要約オッズ比についてのp値関数が青(固定効果モデル)と赤(変量効果モデル)の実線で描かれる(下図)。これは便利だな。
以上の情報は,PRISMAガイドラインについての記述と合わせて,保健学研究共通特講IV/VIIIのテキストに追加しよう……というわけで,新年の会の後にやってしまった。
R-4.2.0リリース
R-4.2のWindows版ではUTF-8をネイティブサポートするというビッグニュース。Rtoolsも変わるそうだ。MSVCRTではなくUCRTに依存しているためWindows10 (1903)以上でない場合,Rより先にUCRTのランタイムをインストールする必要がある。32ビット版もサポートしなくなるそうだ。変化が大きいが,もはや大半のWindowsユーザはWindows 10かWindows 11で,64ビットプロセッサであろうから,良いのではないか。もっとも,内部的な変化も大きいように思うので,最初のうちは慎重にチェックすべきだろうが。(2021.12.20)
R Core Teamのブログ?にあったこの記事のReferenceのDownloadsから,開発中のR-4.2のプレリリース版とRtools42の開発版をダウンロードすることができる。高速デスクトップマシンにインストールしてみたところ,とりあえずR-4.2.0-pre-releaseが無事に起動し,環境変数R_Libsで指定してあるフォルダに既存のR用にインストールしてあるパッケージ群も認識されるようで,fmsbを呼び出してexample(radarchart)をやったら無事に動作した。
しかし,RStudioが動作しなくなってしまった。直接呼び出せばR-4.1.2も動作するのに,RStudioはR-4.2.0 Pre-releaseを呼び出そうとしてエラーが起こり,終了するしかなくなってしまうようだ。ToolsのGlobal OptionのGeneralを呼び出せればR versionの欄で実行ファイル指定できるのだが,Toolsメニューが応答しない。残念だがR-4.2.0 Pre-releaseをアンインストールした。
この原因は,おそらくここで書かれていることで,グラフィックデバイスを提供しているパッケージもすべてdeviceVersionを15にしないと動かないそうなので,RStudioではRの出力をトラップして表示するから,旧来のグラフィックデバイスへの出力をしようとして応答しなくなるということなのだろう。RStudioの開発チームでも既にこの問題は把握していて,解決している感じなので,近々RStudioの新バージョンがリリースされるのではないかと思われる。→RStudioの日本語コードと開発コード名にも書いたが、開発コード幽霊ランなのかエンレイソウなのかわからない2022.02.0+443版で対応済みなので、安心してR-4.2.0リリースを待つことができる。
R-Devel build for Windows (UCRT 64-bit UTF-8)によると,R-4.2では,すべてのパッケージはRtools42を使って再ビルドしなくてはいけないそうだ。まあUTF-8のネイティブ対応は大きな変更だから仕方ないか。とはいえ,大きな変更なのはWindows版だけで,MacOSやLinuxでは既にUTF-8にネイティブ対応していたようだが。
R-devel(たぶん現在だとR-4.2.0)の新しい機能によると,pretty()のboundsオプション付きバージョン.pretty()という関数が実装されるようだ。Windows版の改変が大きいのだが,バンドルされるTcl/Tkも変わると言うことは,RcmdrやEZRにも影響でそうだな。
R-4.2.0(コード名"Vigorous Calisthenics"とは、R-4.1.3のコード名"One Push-Up"、つまり腕立て伏せ1回が掲載されたのと同じ回のPeanutsに入っている言葉で、激しい自重系筋トレを指す。この回のストーリーは、野球帽を被ったチャーリー・ブラウンが、「さあいいかチームのみんな、今日は新しいシーズンの初日だ。みんなで力を合わせて頑張れば凄い年になるぞ」「さて、まずやらなくちゃいけないのは、激しい自重系の筋トレのプログラムを始めることだ」と言ったところで野球用具がたくさん飛んできて、用具に埋もれたチャーリーが「腕立て伏せ1回ならどう?」とつぶやくオチ。R-develで、R-4.2.0ではグラフィックデバイスの扱いや2バイトコードの扱いなど大きく変わるということで話題になり、RtoolsやRStudioも4.2.0対応版にする必要が生じたので、「激しい筋トレ」は適切な比喩と思うし、R-4.1.3が出たときは、「腕立て伏せ1回」はR-4.1.2からの変化が小さいことを意味しているのかと思ったが、実はR-4.2.0への初めの1歩という意味だったのかもしれない)は今日付けでリリースされるはず。Windows版がUTF-8ネイティブ対応になるはずなので、すぐにインストールする予定。
R-4.2.0は無事にリリースされた(R-announceメーリングリストへのPeter Dalgaardさんの投稿)。cloudミラーにバイナリが入ったらダウンロードしよう。
cloudミラーにR-4.2.0とRtools42があったのでダウンロードした。インストールしてみたら無事にできた。Rtools42のインストール先フォルダへのpathは自動的に環境変数に追加されたが、例によってR本体へのpathは自動更新されないので手動更新した。instmyusuallibs.Rを使って自分の常用パッケージをインストールしたところ、これまでと違ってWindows環境でもUTF-8のスクリプトをそのまま実行できて良かったが、パッケージのいくつかはまだ4.2.0対応版がcranミラーに入っていないようで、インストールされなかった。また後日インストールせねばなるまい。
ケンドールの順位相関
裏RjpWikiさんの記事からは度々勉強させていただいているが,Rユーザ,歓喜の涙 -- ケンドールの順位相関係数の計算がPythonの倍速!では,まず使っているRのバージョンに驚いた。M1対応の4.2.0ということは,ここからarm64用のR-develをインストールしているということだな。次いで,RのpcaPPパッケージのケンドールの相関係数がPythonやJuliaより早いという内容自体にも驚いたのだが,pcaPPのGitHubリポジトリを見たら,cor.fk.R()がRでコードされているように見えて一瞬驚いたが,実はその中では.C()でCで書かれたコードをコンパイルしたものを呼び出して計算させていた。まあそれはそうか。これくらい長いとRcppを使うより.C()で呼び出す方が書きやすいのか? どちらも自分でやってみたことはないからわからないが。(追記)自分ではケンドールの順位相関ってほとんど使わないのだが,裏RjpWikiさんは以前も取り上げていたのだなあ。
fmsbにはKendallの順位相関係数τの信頼区間を求める関数が入っていないが、NSM3パッケージにkendall.ci()として実装されている(と、保健学研究共通特講IV/VIIIのテキストにも記載済みである)。この関数のデフォルトはbootstrap=FALSEで、よく知られているフィッシャーのZ変換を使った簡易推定法(リンク先からダウンロードできる論文? には、サンプルサイズが11から24の場合の、0.99から0.40と-0.10から-0.20まで0.01刻みのτについての80%、90%、95%、99%信頼区間を計算するSPSSコードと表も載っているが)だが、bootstrap=TRUEと指定すれば(B=でシミュレーション回数)ブートストラップ推定もできるというすぐれものだが、欠損値対応していないこと(欠損値を含むデータを与えるとエラーになる)、信頼区間だけ表示されて点推定量が表示されないこと、NSM3はかなり大きなパッケージなので、このためだけに使うには大袈裟過ぎる気はしていた。なので、この際、kendall.ci.spss()という関数も作ってみるか……と思ったら、実は、欲しい仕様のものがDescToolsパッケージにKendallTauB()関数として実装済みなのだった。車輪の再発明をする前に気づいて良かった。
R研究集会2021
R AnalyticFlowでローコード分析 by 鈴木了太(株式会社 ef-prime):毎年の進捗。Rユーザー暦20年。R AnalyticFlowはJavaで開発しているマルチOS対応なGUIでRスクリプトを生成し,Rを呼んで実行するソフト。分析フローが作れること,それゆえに簡単に分岐を試せることが特長。(いつの間にかGUIの感じがかなりRStudioに似てきたな。)現在は日本語版と英語版しかないが,内部の仕組みとしては多言語対応していて,世界中からダウンロードはされている。暫くJavaの変化に伴ってLinuxでの動作に問題があったが,問題解決した。2021年8月の3.1.9が最新版だったが,昨日3.2.0がリリースされた。初期バージョンはRスクリプトを書くと(ローコードで)分析フローを作ってくれるソフトだったが,バージョン3からマウス操作だけで(スクリプトを書かずに=ノーコードで)分析フローが作れるようになった。(グラフのプレビューはRStudioより見やすく,むしろjamoviの感じに近い。RStudioとjamoviのいいとこ取りをして分析フロー作成機能を追加したソフトと言えようか?)プロジェクトフォルダ単位での管理がされていて,gitなどの外部ツールとの連携も可能(管理をXMLでやっている)。コード生成の方針として,特定のパッケージに依存せずどこでも動くことを目指してきたが,tidyverseや高度な分析パッケージが使えないのも困るということで,3.2.0では依存パッケージがインストール済みかどうかチェックしてくれて,なければインストールするかを尋ね,ワンクリックでインストールできる。(JREのライセンス問題はどうやってクリアしているんだろう?→チャットで質問した→再配布可能なライセンスのものを使用しているとのこと。ただし今後やり方は変えるかもしれない→奥村先生からご教示いただいたが,OracleのJREも再びフリーで使えるそうだ)ggplot2を使ったデモ。バグフィックスが入った3.2.1を発表中にアップロードしたとのこと。
Rによる福岡市のバス路線網に関する分析 by 橋本芽依(福岡女子大学国際文理学部)/ 藤野友和(福岡女子大学):藤野先生のゼミの4年生の卒業研究。福岡市にある公共交通空白地(バス停からも鉄道駅からも1 km以上離れている場所)が問題。コロナにより運輸業の営業収益が2/3になった問題。バス路線網を企業性と公共性の両方から評価。先行研究(2001)で指標が提案されているので,地域データとGISデータに対してバス停評価指標とバス系統評価指標を算出し,系統再編成を提案するという主旨。企業性について系統ポテンシャル指標,公共性について限定依存人口指標を計算。バス停勢力圏を計算し,その域内の人口を計算(人口はGISに入っているデータを使うのだろうが,移動や自然増減で大きく変わった地域があったら随時追従してくれるのだろうか?),全系統がある場合とある系統を削除した場合との差が限定依存人口指標。ポテンシャルはバス停勢力圏人口×交通発生強度×公共交通輸送分担率で計算。企業性と公共性は,ともに高ければ需要が高く継続可能,企業性だけ高いと営業は可能,ともに低いところは路線廃止や再編成して良い。ただし,企業性が低いが公共性が高い4つの地域は,廃止されたり再編成されると住民が困る。バス停勢力圏の作成方法はボロノイ分割。バス停バッファを500m,完全鉄道バッファを200mとしている。sfパッケージを利用。元データからCrsを設定し平面直角座標系に変換。バッファの作成はst_buffer()関数。ボロノイ分割はst_voronoi()関数。切り抜きはst_difference()関数を使用。(8ヶ月で卒業研究としてここまでやったとは信じられないくらい凄い出来だという谷村さんの感想には同感。福岡女子大ではRでGISを使うという授業をしているので,これが可能になったという藤野さんの説明)
ShinyによるScrapboxの共同編集履歴を分析するアプリケーションの開発 by 河村勇佑(龍谷大学理工学研究科数理情報学専攻):河村さんは修士課程の院生。オンライン授業で複数人で作業し最終的に1つの文書を提出する課題があるが,それだと各人の貢献がわからない。文書作成過程の可視化を目的とし,『RとShinyで作るWebアプリケーション』を参考にしてアプリを開発した。UIとサーバ処理を別々のファイル(ui.Rとserver.R)に記述。先行研究としては,東海大の山本先生の研究室から2本の論文が出ている。Notaが提供するScrapboxを利用。Rのパッケージとしてはtidyverseの他,編集距離(レーベンシュタイン距離)を計算するパッケージ(stringdist?)と日付を扱うlubridateパッケージを利用した。(たしかに,誰がいつどれくらい文書作成にかかわったかがきれいに可視化されていたが,そもそも複数人で文書を作っていくって難しい作業で,量的な寄与だけで評価して良いのか? が気になった→質問したところ,Scrapboxにおけるリンク作成を評価すると質評価ができる可能性があり,今後検討するそうだ)奥村さんが指摘された,レーベンシュタイン距離だと前半と後半のスワップとかコピペで値が大きくなるけれども作業は楽なので,大きな寄与と考えて良いのか? という点も重要。藤野さんからはRで閉じてくれると利用者側にはありがたいという指摘。
RとWebGISを使った環境汚染物質分布の区間推定の表現 by 山川純次(岡山大学大学院自然科学学系):岡山市のPM2.5のクリギングの結果を区間推定を加味してGISで表現するという話。Spread SheetはGoogle,GISはjSTAT MAP(総務省統計局提供)を使用。データは公表されているものをQGISで読み込み,座標系を変換してGeoJSON形式で書き出す。予測グリッドは市区町村別メッシュ・コード。rgdalパッケージを使うとその辺りの操作が大変便利。MacOSを使っているが,Rもパッケージ群もEmacsもESSもすべてソースコードからビルドして使っている。(半ば趣味! だそうだ)推定値を色の種類,CVが大きいほど薄く,QGISでプロットしたのがアイディア。色としてSim Daltonismを使ってCUDを使った。(外部ソフトを使わなくても,Rで表示するならパレットで"Okabe-Ito"を使えばいいのに,と思ったが,QGISやjSTAT MAPでプロットしているのなら仕方ないか?)jSTAT MAPでも界面クロスグラフとして設定すればCVが大きいほど薄くすることは可能。jSTAT MAPはたくさんのデータを含んでいるので,その機能で地形と重ねると,平地ほどPM2.5が高く,山では低濃度である傾向がわかる。谷村さんから,メッシュの座標は中心か左下か? SDではダメでCVでなくてはいけないというのは本当か? と質問。左上で,SDでもダメではないというお答え。中野先生からSDかCVかは目的によるのではないかというコメント。クリギングをするには緯度・経度を直交座標系にしなくてはいけないが,rgdalを使うとそれが簡単だそうだ。
Rによる国際保健医療学分野の二次データ分析 by 谷村晋(三重大学大学院):コロナ禍で調査ができないので,学生が二次データを扱うことが増えた。国別指標のデータベースとして世銀のデータ,個票が使えるデータベースとしてDHSデータの解析について。パッケージとしては世銀のデータについてはWDIとwbstats。DHSについてはrdhs,DHS.rates,prevR。WDIsearch()関数でキーワードから変数名を探せる。ダウンロードはWDI()関数。ところどころデータがない年次とかもある。変数名のattributesとしてグラフのラベルなどに使える文字列も入っている。WDIにはnrow(WDI_data$series)で見ると,17467個の変数が入っていることがわかる。膨大なデータ。DHSは調査ユニットの位置情報も調査データそのものの使用申請とは別に申請する必要があるが使える。DHSのデータ入力はCSProで行われているが,4つの形式のフラットファイルでダウンロードできる(どの形式が良いかはわからない)。RData形式はない。rdhsパッケージを使えば,DHS APIを通じてDHS指標一覧にアクセスでき,必要なデータセットをダウンロードしてRの中で読み込み,メタデータと関連付けて分析できる。surveyIDsがわかったらdhs_datasets()関数でデータをダウンロード。その後にget_datasets()でダウンロードできるはずだが,まだ成功していないとのこと。データを直接ダウンロードする方が早い。DHSデータからTFR,GFR,ASFR,NNMR,PNNMR,IMRなどを算出してくれるパッケージがDHS.rates。foreignパッケージのread.dta()でDHS.ratesに入っているStata形式のデータを読み,fert()関数を使うとTFRの値をSEや信頼区間付きで推定できる(信頼区間はジャックナイフで推定)。chmort()で子どもの死亡率の指標がIMRやU5MRなどいくつか計算できるが,CMRが何を意味するのか不明とのこと(→計算式などの詳細は,PLoS ONEの論文に載っていた)。prevRは(DHSからSPSS形式のデータをダウンロードしないと使えないが)クリギング法による空間補完を行うことができる。(データの質について問題が出ることはないか尋ねてみたが,DHSではこれまでそういう問題を聞いたことはないとのこと)
Rによる遺伝子ネットワーク解析 by 樋口千洋(国立研究開発法人 医薬基盤研究所):樋口さんが毎年発表されているオミックス解析の続き。ゲノミクスではバリアント数×遺伝子型を解析。形質に対する線形回帰のp値を算出。トランスクリプトミクスではRNA×サンプル数。プロテオミクスではたんぱく質数×サンプル数だが,AlphaFold2という画期的なソフトができたのでこれまでのいろいろな問題が解決した。メタボロミクスではm/z×サンプル数×保持時間で,既に機械学習をこれに適用する話を報告した。実はゲノムでもDNAが修飾されるかどうかで発現するかどうか変わるので,トランスクリプトミクス同様な考察もできるはず。トランスクリプトミクスにおいて,DNAのイントロン部分から転写される短鎖RNAであるmiRNAが他のRNAに干渉することが最近わかってきて,重要な働きをしているので分析対象となっている。RNA発現データはNCBI GEO(発現データセットのリポジトリ)で検索取得可能。次世代シークエンサのデータは生データで提供されていることが多く,利用者が正規化データに変換する必要がある。Rでの読み込みはread.csv()でOK。欠損値が含まれている場合と,欠損値がNULLという文字列で表記されている場合があり,それらは行単位か列単位で削除する。R-4.*での変更が多少影響あり,昔作ったプログラムがそのままでは動かないことがある。遺伝子ネットワーク解析をする際,一般に因果推論をするのにベイジアンネットワークが使えるが,処理速度やサンプルサイズが問題になることがある。相互情報量によるマルコフネットワークの方が早い。FSL (Forest Structure Learning)はマルコフネットワークの1つ。BNSLパッケージで鈴木譲先生考案の相互情報量推定ができる。ARACNeだとぐちゃぐちゃになるようなデータでもFSLだとすっきりする例。
He swung and a miss! Clustering swinging strikes - The case of Shohei Ohtani by 服部恒太(徳島大学):15年前くらいからRを触るようになった。昨年は米国大統領選のヒスパニックの投票に影響する要因と関西弁のある表現の拡散の話をした。今年はセイバーメトリックスを扱っている学生がいて,空振りの研究をするということで,大谷さんのデータを分析。baseballrパッケージを利用。投手の左右と球種別の棒グラフでみると,左右どちらでもフォーシームで一番空振りしていた。ストライクゾーンのどこで空振りしているかを空間統計手法で図示すると,真ん中高めと低めで空振りが多いように見える。plate_x,plate_z,release_speed,release_pos_x,release_pos_y,release_spin_rate,pfx_x,pfx_z,vx0,vy0,vz0,ax,ay,azを分析。2018-21の4年分のデータを利用。PCAで4つの主成分で80%説明ついた。パラレル分析でも4主成分。PC1とPC3は球種絡み,PC2とPC4は投手の利き手絡みらしい。HDBSCAN(Rではcranにあるdbscanパッケージ)でinternalとexternalのクラスターを分析。クラスター1は右投手のインロー,クラスター2は左投手のアウトローへのスライダー,クラスター3は左投手のインハイへの速球,クラスター4は右投手の真ん中低めボール気味にくるチェンジアップ,クラスター5は右投手の真ん中高めボール気味にくる速球,それぞれカウントごとの傾向が違う。(4年分まとめた分析だったが,かなり打ち方を変えている気がするので,空振り傾向も年ごとに変化していないか?) チャットに方言研究の協力依頼があったが,これはリンクしていいかどうかわからないのでURLはメモしない。
R による財務ビッグデータのラングリング再考 by 地道正行(関西学院大学商学部):1999年からSを使い始めた数理統計学者。4年前に,世界の企業2000万社超の財務情報の解析を志した。試しに100万社。ファイルサイズ4 GB。read.table()で読んだら,読めたように思われたがデータがおかしくなった。500万行を超えるとおかしい。100万行ずつ読んでつなげればOK。readrパッケージのread_tsv()で問題解決。ギガ単位のデータを正しくラングリングするのに適したハードウェア,OS,Rの関数を探索。約290万行,2900万行,2億9000万行で,100列弱の3つのCSVファイルを使って検討。MacBook Pro,iMac Pro,Mac mini 2020,DellのPrecisionサーバーでUbuntu,Core i7のWindowsマシン,東大のFENNEL,mdxを比較。Rパッケージはbase,data.table,readr,feather,arrow,vroomを比較。Windows 10では290万行ファイルではvroom::vroomが最速だが,2900万行ファイルだとreadrもvroomもこける(実メモリ64 GBなのに)。data.table::read_csvは安定して読める。MacOSやLinuxではvroomもOK。parquet形式にシリアライズしたファイルはarrow::read_parquetで早く読める。(質疑では,メモリ上限による制限の話―この辺の話か?―で盛り上がった)
OSSベースでのRパッケージ開発のすすめ by 瓜生真也(徳島大学デザイン型AI教育研究センター):これまでパッケージを9つcranに登録している。いくつか登録後削除しているものもある。登録していないものも含めれば50くらいのパッケージを開発した。石田先生,服部先生もいるし,ホクソエムの方が客員准教授になっているので,Tokushima.R企画中。OSSでのパッケージ開発を勧める理由は(1)コミュニティ・研究への貢献,(2)問題解決・機能追加の速度を向上,(3)開発意欲の維持。個人開発だと孤独で意欲が保てないかもしれない。(ここで「OSSベースでの」というのは,いわゆるバザール方式をさしているようだ。cranに載っていれば個人開発でもほぼソース公開だと思うが)これまで瓜生さんが作ったパッケージのうちいくつか紹介。jpmeshは標準地域メッシュを扱うパッケージ。fgdrは国土地理院が提供する基盤地図情報の処理をするパッケージ。zipanguは日本語まわり。Nipponパッケージが無くなってしまったので。OSSとして多くの開発協力者。jmastatsは気象庁データを取得・整形して提供するパッケージ。気象庁サーバへの負荷が増えることを懸念してcranには入れていない。OSSとして開発したことにより,新しい技術に触れたり協力者が得られたりしたのは大きなメリット。JOSS,ZenodoはGitHubと連携,rOpenSciも含め,パッケージが研究業績として評価される可能性あり。今後パッケージ開発をする人をTokushima.Rでサポートしていきたい。(凄い!)開発コミュニティのうち,パッケージメインテナに全員を入れるわけではないが,新機能を追加した人などは入れるそうだ。
Gneralized Estimating Equation (GEE)
Rでは,https://cran.ms.unimelb.edu.au/web/views/SocialSciences.html(注:CRANにTask Viewといって,分野別にどのようなパッケージが使えるかについてのガイドがある)によると,gee,geepack,multgeeなどのパッケージでできるようです。
geepackはhttps://cran.ms.unimelb.edu.au/web/packages/geepack/vignettes/geepack-manual.pdfというマニュアルがあります。multgeeもhttps://cran.ms.unimelb.edu.au/web/packages/multgee/vignettes/multgee_vignette.pdfに丁寧な説明があります。
TaskViewには入っていませんでしたが,mgee2というパッケージもあります。
これらの文書や,http://bin.t.u-tokyo.ac.jp/summercamp2017/file/2-7.pdfや,https://mstour.hatenablog.com/entry/2021/03/12/205654を見た感じだと,必ずしも欠測データ処理に向いた手法というわけではないようで,むしろポイントは,パネルデータのようなものに対して当然想定される説明変数間の相関を積極的にモデルに入れるというところだと思います。
GEEについて大阪市立大の新谷歩先生(注:週刊医学界新聞で「今日から使える医療統計学講座」を連載されていたヴァンダービルト大学から阪大に移られたのは知っていたが,その後大阪市立大に異動されたとは知らなかった)がYouTubeにいくつも説明動画をあげていました。学生へのインストラクションとしては,まずこれを見てもらうのが良いのでは?
- https://www.youtube.com/watch?v=1z2elgspV-g
- https://www.youtube.com/watch?v=T73UwC_kQTw
- https://www.youtube.com/watch?v=5dx_d_K0kWs
- https://www.youtube.com/watch?v=89LghOd0X2Q
- https://www.youtube.com/watch?v=Hr0MbvJQTMI
fmsb-0.7.2
2020年の国勢調査人口が公表されたので,少なくともfmsbパッケージのJpopとJpoplは更新できることに気づき,fmsbを0.7.2に更新してCRANにアップロードしてみた。単なるデータ追加なので,たぶん問題なく承認されるだろう……と思っていたが,ドキュメント(Rd)にデッドリンクがあると通らないことを忘れていた。データソースのURLが消えていたり変わっていたりするので,それを全部訂正しないと。(2021.12.11)
URLを全部直してから再投稿したのだが,Windowsでは通るけれどもDebianでエラーが出ているというコメントが来て困っている。Kurt Hornikさんの環境でのエラーログが提示されているのだが,2バイトコード絡みらしく,fmsbでは使っていないので,こちらでは対処できない気がする。update.packages(ask=FALSE, checkBuilt=TRUE)をやったら,ソースからコンパイルするときに似たようなエラー(警告?)が表示されたので,もしかするとgccの問題なのではなかろうか?(2021.12.8)
Uwe Liggesさんからメールが来ていて,URLの変更については直す必要があるが,Debianのエラーは無視して良いそうだ。一安心。(2021.12.9)
fmsbパッケージのradarchart()関数の使い方を紹介してくださった記事、ありがたい。fmsbパッケージの中ではたぶん一番よく使われる関数で、最近だと、Yu Y et al. "Immune Checkpoint Gene Expression Profiling Identifies Programmed Cell Death Ligand-1 Centered Immunologic Subtypes of Oral and Squamous Cell Carcinoma With Favorable Survival"(Frontiers in Medicine, 2022.1.20)やPohl S et al. "Age and Appearance Shape Behavioral Responses of Phasmids in a Dynamic Environment"(Frontiers in Ecology and Evolution, 2022.1.21)といった用例がある。
CRANにtaylorというパッケージがあって、Taylor Swiftの全楽曲データが入っているらしい。それを使ってデータ解析をしている記事で拙作fmsbパッケージのradarchart()関数が使われていた。
R-4.1.2リリース
R-4.1.2(コード名"Bird Hippie")が予定通り11月1日付けでリリースされたそうだ。Bird Hippie初出の回をtweetしたら,Core TeamのPeter Dalgaard大先生からご返事いただき,ハロウィーンネタの1967年11月1日の回を想定していたことをお教えいただいた。もしかすると,これまでのコードネームもリリース日付の回のPeanutsからネタをとっていたのか?
『計量分析One Point イベント・ヒストリー分析』
共立出版から,Paul D. Allison(著),福田亘孝(訳)『計量分析One Point イベント・ヒストリー分析』,共立出版,ISBX978-4-320-11411-1をご恵贈いただいた(ありがとうございます)。U Penn名誉教授のPaul D. Allisonによる"Event History and Survival Analysis 2nd Ed." (2014)の全訳に訳者補遺としてRで解析する方法の解説が追加されていて,コンパクトで読みやすい。原著の分析コードはSASとStataで提供されていて,それもwebからすべてダウンロードできるそうだが,訳者補遺のRコードも共立出版のウェブサイトからダウンロードできる。Stataのdta形式のデータを読み込むためにhavenパッケージのread_dta()を使い(古典的にはforeignパッケージのread.dta()だが),一部データ前処理のためにtibbleやパイプ演算子%>%を使っているけれども,gridグラフィクスというかggplot2を使っていないので馴染みやすく感じた。
『統計的因果推論の理論と実装:潜在的結果変数と欠測データ』
高橋将宜さんがWonderful Rの第5巻として出版予定の本は大変興味深く役に立ちそうな内容。買わねば。高橋さんのtweetからすると,Wonderful Rシリーズは査読があるんだな。扱われるテーマが広いので,もし石田さんが全部査読しているなら大変だろうと思うが,まあ普通に考えたら,巻ごとに近い分野の人に振っているんだろうなあ。(2021.9.7)
アマゾンで予約購入した、高橋将宜『統計的因果推論の理論と実装:潜在的結果変数と欠測データ』共立出版、ISBN:978-4-320-11245-2が届いていた。前著『欠測データ処理』が名著だったので本書にも期待して購入したが、傾向スコア、操作変数法、回帰不連続デザインについて、Rのコードを動かしながら自習できる(データやコードが著者のGitHubページで提供されている)という点で、多くの人から待ち望まれていた内容だと思うし、院生にも薦めたい。(2022.2.10)
R-4.1.1リリース
R-4.1.1がリリースされたというPeter Dalgaard師のtweet。開発コード名はKick Thingsで,出典はたぶんこれ。サッカーボールを蹴り続けるルーシーに対して,チャーリーがあれこれと理由を推測して言うのだけれども,最後に「私はただ物を蹴るのが好きなの!」と言い放つルーシーというオチ。[Rd]で流れてきた情報を見ると,結構色々変わっているなあ。qnbinom()が極端な値についても正しい結果を返すようになったとか。(2021.8.10)
R Journalの13巻1号が公開されたというメールがR-announceメーリングリストに流れたのでチェック。新しいパッケージを紹介するたくさんの論文に加え(gtsummaryパッケージは便利そうだと思ったが,説明がパイプ記法なのだよなあ),R-4.0からR-4.1への変更点まとめとcranの状況変化がわかりやすい。登録パッケージ数が指数増加まではいかないが,それに近いペースで増えていることがわかる。
Windowsユーザなので知らなかったが,裏RjpWikiさんによると,MacOSだとgmpパッケージとRmpfrパッケージはソースインストールにしないと動かないことがあるそうだ。
fmsb-0.7.1
去年の9月に確定数が発表済みなのに,2019年の人口動態統計をfmsbに入れてアップデートするのを忘れていたことに気づいた。作業せねば。
fmsb-0.7.1のドキュメントに書いてあるURLの中にinvalidなものがいくつかあるので修正して再投稿するように,という指示メールが届いた。ドキュメント全部を一々チェックし直していなかったので,データソースにしたURLなど,既に消滅してしまっている場合もあり,その場合は消すべきということらしい。もちろん移転したりhttpがhttpsになっただけなら,新しいvalidなURLに直さねばならない。面倒だが仕方が無い。しかし,今日は講義の他,某書類をオンライン提出しなくてはいけないので,それが終わってからだな。
というわけで某書類のオンライン提出を済ませてからfmsb-0.7.1のURLを修正してbuildし直したところで18:00になった。
いろいろな論文で使われているfmsbパッケージ(主にradarchart()だが)には,日本の人口データをいくつか収載しているので,定期的なアップデートをするべきなのだが,2019年の人口動態統計確定数が随分前に出ていたのを忘れていた。追加してfmsb-0.7.1としたところ,0.7.0では存在した情報源がいくつもデッドリンクになっていて,その修正が大変だったが,やっとcranに載った。例えば,library(fmsb); plot(TFR ~ YEAR, data=Jvital, type="l")とすると,2018年までのデータを使った場合と最後のところの印象が随分変わる。(2021.5.11)
保健学研究共通特講IV, VIIIの今日のトピックは2つの量的な変数間の関連で,相関と回帰(の一部)について説明した。スピアマンの順位相関係数の信頼区間を求めるところで,fmsbパッケージに実装したspearman.ci.sas(...)の結果がcor.test(..., method="spearman")と合わずに慌てたが,後でチェックしたら,spearman.ci.sas()の関数定義の中では欠損値処理していないことを忘れていた。getS3method("cor.test", "default")とすればわかるようにcor.test()は関数定義内部で欠損値をペア単位で除去しているから欠損値を含むデータを与えても正しく動作するが,cor.test(rank(x), rank(y))とかspearman.ci.sas(x, y)とやってしまうと,xとyが異なるケースで欠損を含んでいると当然結果が狂う。fmsbパッケージの次回更新のときに直そうと思うが,とりあえずテキストに注記して,テキストのrevisionを1.0.1とした。
Breslow-Day検定
RではDescToolsパッケージにBreslowDayTest()という関数が実装されている。すべてのOR=1という特殊な帰無仮説については,パッケージを使わなくてもmantelhaen.test()で実行可能だが。BreslowDayTest()は,リンク先の説明を読む限りオッズ比がどの層でも均質であるという帰無仮説を検定するようだ。この論文はオッズ比ではなく罹患率比の比較なので,さらに調べてみたところ,epiRパッケージのepi.2by2()でオプションとしてmethod="cohort.count"とhomogeneity="breslow.day"を指定すれば,罹患率比についてBreslow-Day検定をしてくれるという情報があったのだが,1.0-04版でバグフィックスとしてhomogeneity=オプションが削除されていた。かつ,罹患率比はmethod="cohort.time"を使わなくては計算してくれず,そのときは層間比較をしてくれないという仕様に変わっていた。この論文で使われているのはリスクではなく罹患率で,分母としては観察人年しか与えられていないので,期首人口を計算できない(観察期間が各年最大153日なので,それで割ってみたり,さらに365.24をかけてみたりしたが,整数にならないということは,途中参加や脱落があるのだろう)。だからリスク比やその均質性は計算できないのだが,観察人年から急性心筋梗塞罹患数を引いた値を非罹患数として無理矢理計算してみると,論文掲載の値と概ね一致した(Rコード)。が,これでいいとは思われないのだがなあ。Breslow and Dayの本のうちコホート研究の分析を扱っているVolume 2はpdfが公開されていたので読んでみようと思うが,ざっと見ただけではどこに載っているのかわからなかった。
昨日ダウンロードしたコホート研究を扱っているVolume 2でなくても,たぶん層間での比の比較という点ではVolume 1に書かれている内容を読めばBreslow and Dayの均質性の検定の考え方はわかると思い,IARCのサイトを再び見ていたら,多数の本が無料でpdfやepubをダウンロードできるという素晴らしい事実に気づいた。例えば,IARC Scientific Publication No. 161 "Air Pollution and Cancer" Edited by Straif K, Cohen A, Samet J (2013)も全文読めるし,IARC Scientific Publication No. 168 "Reducing Social Inequalities in Cancer: Evidence and Priorities for Research" Edited by Vaccarella S, Lortet-Tieulent J, Saracci R, Conway DI, Straif K, Wild CP (2019)も全文読める。1980年代の出版物は紙の本だけの場合も多いが,Breslow and Dayは,有り難いことに症例対照研究を扱っているVolume 1もここからpdfをダウンロードできた。"Test for homogeneity of the odds ratio"は142ページから書かれていて,どの層でも同じ比が観察されると仮定したときに期待される各層の曝露あり疾病ありの人数を,観察されているその人数から引いて二乗し,各層の分散で割った値をすべての層について合計したカイ二乗値が層数マイナス1のカイ二乗分布に従うとして検定する,カイ二乗適合度検定のようなロジックのようだ。これをリスクでも率でも同じように援用すれば良いのだとしたら,たぶん罹患率比でも同じ式で問題ないのかもしれない。まだ若干釈然としないが,とりあえずこの問題追求はここまでにしよう。他にやらなくてはいけないことが多々あるし。
equatiomaticパッケージ
凄いパッケージが出ているのを知った。equatiomaticという(リンク先でVignettesを見れば使い方はだいたいわかる)。Rでlm()とかglm()ならまだいいが,lme4やlmerTestパッケージのlmer()関数で混合モデルやプロビットモデルを当てはめるコードを書いて解析した場合,その式を論文のmethodに書くのが面倒で,間違う場合も少なくない。これを使うと,lmer()で使ったモデルが,そのままTeXのコードになる。LaTeXで論文を書いている人はそのまま取り込めるし,WordやLibreOfficeで論文を書いている人でも,(1)ヘッダをつけてpandocでWordのdocxやWriterのodtに変換するとか,(2)MS Office+IguanaTexを使ってWordに取り込むとか,(3)LibreOffice+TexMaths拡張を使ってLibreOffice WriterやImpressに取り込むなどすれば,LaTeXがわからなくても使えてしまう。forecastパッケージのArima()関数にも使えるようだ。これは私的常用パッケージ入りだな。
『Rで計量政治学入門』
Rで計量政治学入門は素晴らしい資料だと思う。とくにIIIデータの収集が役に立ちそう。そこからリンクされているrtweetパッケージの使い方の記事も素晴らしい(cranのrtweetのページにはvignetteもいくつかある)。infodemic対策の準備として,今日の「新規感染者数」というトレンド入りしたtwitter検索語でのスクレイピングをやっておかねば。何日も前に出た論文で,専門家はそのとき軽くディスカッションしているのだけれども,数日経ってテレビメディアが取り上げたときに,おそらく戦略的に体系的になされているものも含めて,どのように(相互作用しながら)メッセージがついていくのかを検討するのに適した材料な気がする。search_tweets()関数とsave_as_csv()関数だけでも何とかなるが,この文書とか開発者のGitHubページも参考になりそう。
『データ分析のためのデータ可視化入門』
キーラン・ヒーリー(著),瓜生真也,江口哲史,三村喬生(訳)『データ分析のためのデータ可視化入門』講談社,ISBX978-4-06-516404-4をご恵贈いただいた。tidyverseを使ったデータからの作図のガイドブックだが,なぜデータを見るのか,悪いグラフのどこが悪いのか(3Dグラフの問題点やアスペクト比の違いが与える印象の違いなど)に続く,1.3の知覚とデータ可視化,が面白かった。2章からは実践だが,RStudioでRmarkdownを使ってやっていくスタイルだが,ggplot超入門みたいなところから始まるのでとっつきやすいかもしれない。8.5.3「円グラフは使っちゃダメ」は,base Rのpie()のヘルプにも書かれているとtweetしたばかりだったので良いタイミングだった。
欠損値処理
欠損値処理の質問に答えるため,mvnmleパッケージについて調べてみた結果わかったこと(なお,無料で読める日本語による欠損値処理の説明としては,覚え書きとのことだが,村山航 (2011) 欠損データ分析(missing data analysis) ―完全情報最尤推定法と多重代入法―がわかりやすいと思う。英語だとcranのMissing DataのTask Viewやfinalfitというパッケージのvignetteがコンパクトでわかりやすい)。mvnmleは2012年頃にlme4の開発者であるDouglas Batesの協力を得てKevin Grossという方が開発しメンテナンスしていたが,既にメンテナンスを止めていて,cranではORPHANEDという扱いで,去年5月に削除済み。しかしインストールはGithubから可能で,devtoolsパッケージがインストール済みなら,
library(devtools)
install_github("cran/mvnmle")
とすればインストールできる。ちなみにKevin Grossは2015年からテニュア付きの教授職を得ているが,webサイトにはmvnmleのことはカケラも載っていないのでもうメンテする気はないのだろう。LittleMCAR()についてはこの説明によると,BaylorEdPsychパッケージにも入っているようだが,このパッケージも既にcranから削除されている。LittleのMCAR検定については,メンテされていないパッケージを使うより,本当は,Little RJA (1988) A test of missing completely at random for multivariate data with missing values. Journal of the American Statistical Association, 83: 1198-1202.を読んで自分でコーディングした方が良いと思う。Stataには実装されているので,Rに現行のものがないとは思えないのだが……と思ってTask Viewを見直してみたら,RBtestパッケージでMCAR性の検定ができるそうだが,典拠となる文献が書かれていないし,回帰に基づく方法ということでLittleの方法とは違うようだ。なお,質問者が陥ったというエラーは,たぶんここに書かれている例みたいに,すべて欠損な変数が入っているのではないかと推察している。
JSSに載っていた論文によると,JointAIというパッケージで欠損値に対する多重代入法ができるようだ。例としてNHANESの個人レベルのデータが使われている。この本のChapter 6でも解説されている。開発はこのGitHubページ。
Juliaとの連携
裏RjpWikiさんのJuliaとRを行ったり来たりという記事に興味を引かれたので,ここからWindows版64ビット環境をダウンロードしてE:\Juliaにインストールしてみた。しかし,このアイコンを起動してもadd RCallなどできないので(終了はunix系OSのシェルなどと同じくCtrl-Dでできた)調べてみた。この情報を参考にして,まずJuliaの起動アイコンのプロパティで作業フォルダをE:\Julia\binにした……が,それだけではPkg.init()も通らないなあ。次に環境変数を設定してみたが,やはりUndefVarErrorが出るので,さらに検索したらこのページに1.0系では"]"キーを押してPkgモードに入れば良いと書かれていて問題解決した。よく見たら起動時のメッセージに"]?" for Pkg helpと書かれていて,"]"を押せば良いことはそれだけでわかったはずなので,これはぼくが間抜けだった。
で,早速Pkgモードに入って"add RCall"をしてみたのだが,これ時間掛かるなあ。お,できた。using RCallをするにはPkgモードから抜けなくてはいけないのだが,抜け方がわからないなあ……MacではDeleteキーと書かれていたので,もしかしたら? と思って試したらWindowsではBackSpaceキーで良かった。つまりCtrl-hを送れば戻れる(あとで見つけたが,ここに書かれている説明がコンパクトにまとまっていて便利)。using RCall以降は裏RjpWikiさんの記事に書かれている通り,JuliaとRを行ったり来たりは簡単だった。オーバーヘッドがあるのか,もちろんR"*****"をJuliaで実行するよりRで直接*****を実行する方が速いが。obj = rcopy()でRの実行結果をJuliaのオブジェクトに取り込めるのは便利だ。
昔は専らRについてマニアックな考察を展開されていた裏RjpWikiさんが,一時Pythonに手を出していたが,最近はJuliaにハマっているようで,200以上のコード実装例を書かれている。チュートリアルがあるなら,手を出してみようかなあという気になってきている。
R研究集会2020
中野先生がチャットにTidyverseSkepticを貼り付けられた。最初のところだけ斜め読みした感じでは,便利とか見やすいといって薦められることが多いtidyverseは,R初学者にとっては,むしろ学習の壁になっているという主旨のように思われる。ぼくもそう思う。
最初の演題は統数研の川崎さん「RS-Decomp」。R-Shiny-Decompの略。今日が本邦初公開とのこと。Decompとは一変量非定常時系列データを複数の要素時系列に分解するソフトとのこと。方法論はKitagawa G (1981) J Time Ser Anal, 2(2): 103-116.で,Fortran77のソースコードは統数研のTIMSAC 84 Part1にも載っている。RS-Decompは,そのRへの移植ということか? COVID-19のdailyの新規確定患者報告数データにも使えそうだが,季節調整が本来の主目的とのことだから,むしろ食中毒とか季節性インフルエンザの方が適しているのかも。yn=tn+sn+wn,wn~N(0, σ^2)と考えてynをトレンド成分tn,季節成分sn,不規則変動(ノイズ)wnに分解したいが,サンプルサイズTに対してパラメータが2T+1個あるので不適切問題。そこで時間方向に自然な制約として,平滑化事前分布を導入することで,実効パラメータ数を減らす。なるほど。DECOMP(1985)は分解要素の定常AR成分を含むこと,曜日効果にも対応していること,状態空間モデルゆえ欠測・予測に統一的対応がとれること,非定常成分の状態初期分散を厳密に初期化すること,が特徴。Web Decomp(佐藤整尚先生)が偉大な先駆者。decompをDLL化してS ver. 4からコールし,それをウェブのフロントエンドから操作するものだった。2006年にuseR! 2006で佐藤さん自身が発表している。が,最近,プラットフォームだったWindows 2008 Serverがサポート終了したので終了した。統数研はcranにtimsacパッケージを維持しているが,timsacに入っているdecomp関数のメンテをRS-Decompに反映することにした。統数研の時系列モデルとしては,2020年12月15日に北川源四郎『Rによる時系列モデリング入門』(TSSSパッケージの使い方説明を含む)が出たとのこと。操作デモを見せていただいたが,使いやすそうだった。ちなみに,北川先生のFortranコードは,以前は1万円くらいで売られていたが,今は公開されていて,webからダウンロードできるとのこと。
2番目の演題は徳島大学の学生さんの発表。服部さんが指導教員。USの大統領選挙で投票に影響した要因の分析。先行研究として,さまざまな地理的要因,人種(黒人は民主党支持,白人は地域によって傾向が異なる,Teigen et al. 2017)などの影響が指摘されてきた。今回の分析ではヒスパニックに注目。先行研究ではヒスパニックは民主党支持が多い,あるいは出身国によって異なる,違法移民への見方が世代や出身国によって異なるといった指摘がある。しかし,2004年の大統領選でヒスパニックの投票傾向を左右したのはモラルやテロ対策だった(Abrajano, 2008)。当時は銃乱射,移民への排他的政策,地球温暖化などが問題だった。2016年も社会不安はあったので同じ傾向ある? CCESというサイトから入手した2016年大統領選に参加した6万人のサンプルデータ(オープンデータ)をRで分析した(ただし,ヒスパニック,かつ欠損のないデータだけ使ったので,)。年齢など,さまざまな属性や要因も含まれている。銃規制への態度などは複数の賛成/反対質問への賛成の数を変数とした。目的変数は,トランプとヒラリーのどちらに投票したかの二値で,ロジスティック回帰(ステップワイズの変数選択)。人口密度データも別途作って結合した。結果としては,銃規制賛成なほど,移民に寛容なほど,環境保護傾向があるほど,人種差別の存在を認識しているほど,ヒラリーを支持していた。その他では,保守主義(どういう質問によるのか?)の人がトランプ支持,独身と離別した人はヒラリー支持。トランプ支持者が反知性主義という話は良くあるが,学歴は? と尋ねてみたら,ステップワイズの選択で残らなかったとのこと(疫学ではステップワイズは薦めないが,まあそれはそれとして)。ただし,指導教員の服部さんから補足コメントがあって,全人種でやると学歴は効いていて,大学・大学院卒が民主党支持だったとのこと。
3番目の演題は総務省統計研究研修所と独立行政法人統計センターの方々による,e-Stat APIを用いたスクレイピングについて。使い方の説明と,家計調査データを使った使用例。コードはここから入手できる。ただし,動かすには,e-Statへのユーザ登録をしてアプリケーションIDを発行して貰う必要がある。家計調査は全国約9000世帯に家計簿を付けて貰ったデータ。毎月調査だが,今回のデモでは,二人以上世帯の年次データを取得して分析。リクエストURLの中にデータID(statsDataId)が含まれている。RでのスクレイピングにはRCurlパッケージとjsonliteパッケージを用いる。RCurlパッケージのgetURL()関数にユーザIDやデータIDを含むURL文字列を与えでデータを取得し,jsonliteのfromJSON()関数でデータとして使えるリストに変換する。GET_STATS_DATA$STATISTICAL_DATAというリスト要素が,実際のデータを含むリストになっている。そのDATA_INF$VALUEがデータフレーム。属性コード6変数と統計数値は文字列として1変数。CLASS_INF$CLASS_OBJ$CLASSが属性情報の内容に対応。それらの情報を使って続きのデータを取り込まなくてはいけないし,ここから手作業でデータを取り出すのはメチャクチャ面倒くさいと思うので,むしろ普通の二次元表形式のデータフレーム(またはそのリスト)に自動変換する関数を書いてしまう方が良さそう。あ,もしかすると,この発表は,最後はそういう関数を作りましたということに帰着するのか? と思ったが,そうでもなかった。こうやって抽出したデータをクラスタリングしたという報告が,エストレーラに掲載予定とのこと。@は変数の頭に使えない文字というより,S4オブジェクトのスロットを意味するオペレータだよなあ。
4番目はRとPythonの比較。これ,最近裏RjpWikiさんが連投しているネタと近いのか? 2015年useR!の思い出。スパース推定の本を書くのに際して,先にRコードを書いて,Python版も作る必要があったので移植したら3ヶ月掛かった。パッケージにすると中身を見なくなるのでスクラッチで書いた。コードはここで公開されている。LASSO推定などのスパース推定について,Stanford大学のFriedmanらが開発したglmnetやglassoはRパッケージとしてcranで公開されているが,Python版はLinuxでしか動かない。scikit-learnというボランティア開発のものはあるが,動作が本家と違うので怖くて使えないとのこと。RとPythonの比較。
5番目は谷村さん。共有でなくカメラでプレゼン。ObsStudioでも使っている? (いま気づいたが,たぶんこの方がZoomへの負荷は小さいので,木曜の全学の講義で,スライド転換がうまく動かなかったときに,これをやれば良かった!)Rでのカラーユニバーサルデザイン対応の話。最初は色覚異常についての説明。川端裕人『「色のふしぎ」と不思議な社会:2020年代の「色覚」原論』筑摩書房に載っているような話。PAとかの6パタンの説明は,谷村さんが示した表は大変わかりやすかった。色覚バリアフリーの作図ポイントとして,(1)配色をsafe colorに,(2)ハッチング併用,(3)境界線,(4)色区別がないと判別できない線を使わない,(5)色区別がないと判別できない凡例は使わない,(6)彩色面積を大きくする。まずはダメなグラフの例。rainbowカラーで境界線無しの帯グラフ(Rで普通に作ると境界線は付くが)とか,matplot()でtype="l"でcolだけで判別する折れ線グラフとか。barplot()では彩色とハッチングが両立しないのでadd=TRUEを使って重ね描きする。折れ線グラフはltyを使って線種も変える。色自体も変えられる。shadesパッケージにはdichromat()という関数があり,P型,D型,T型の人にどう見えるかがわかる。DescToolsパッケージのColToGray()関数ではグレースケールに変換できる(正解はないが)。monochromacyの人にもこれで良いかは不明。ではどういう色を使えばカラーユニバーサルなのか? というと,safe colorsetsを提供するパッケージが複数ある。今回はcolorBlindnessパッケージを紹介。Wong (2011)のcolorsetsを収録している。displayAvailablePalette(color="ivory")で使えるパレットが一覧でき,displayALlColors(safeColors, color="ivory")などとするとパレットが表示される。safe colorに色置換することもできる。colorBlindnessパッケージのreplacePlotColor()関数が使える。ただし引数がgrobクラスオブジェクトでなければならず,baseグラフィックではダメで,base2grobパッケージなどでgrobクラスオブジェクトに変換し,gridグラフィクスで描画しなくてはいけない。が,それだとまともに機能しているとは思えない結果になったので,最初からsafe colorのパレットを使う方が良い。palsパッケージのpal.safe()など,今日紹介された他にもいろいろあるなあ。R-4.0.0以降での変化という話があったが,palette("Okabe-Ito")が,色覚異常対応のために導入されたものらしい。従来のカラーパレットに戻すにはpalette("R3")で良い。palette.pals()で使えるパレットの一覧が出てくる。barplot(1:7, col=1:7)などとすればパレットが切り替わったことは確認できる。
6番目はRAnalyticFlowで有名な鈴木了太さんの発表で,クラウド&リモート時代のデータ分析環境,という話題。COVID-19の影響でリモートでの業務遂行が重要になった中,データ分析環境をどう拡張・移行するか。スタンドアロン vs オンプレミス(イントラネット) vs クラウド。スタンドアロンでは,データはプレーンテキストかExcelなどのファイルを,R(+RStudio)やExcel,Access,SQLiteなどで,1台のコンピュータの中で完結。オンプレミスではデータは共有サーバにあり,分析もサーバにリモートデスクトップやsshで接続して実行。クラウドでは,データはクラウドストレージ上,分析用仮想マシンや,分析用のクラウドサービスを使う。リモートで在宅勤務の時はどうするか? (1)オンプレミスネットワークにVPN接続,(2)データや分析環境をクラウドに移行,(3)クライアントPCのリソースを活用。(3)では,バージョン管理システム(Gitなど: GitHubやGitLabやSourcetree)を用いてデータやスクリプトを管理し,原則としてローカルで作業し,データ送受信は作業の合間にすること,PCのストレージをキャッシュとして使い(OneDriveやGoogle Driveなど),コンテナ型の仮想環境(Dockerなど)を活用することが考えられる。
7番目は英語の発音がメチャクチャ格好良い服部さん。言語の語彙がどう拡散していくか("lexical diffusion"というそうだ)をTwitterを使って関西圏で分析したという話。従来はフィールドワークをして生の言語を集めていたが,大変な仕事だった。関西弁の新しい否定の助詞「やん」の拡散を調べた。元々の否定の助詞としては「へん」と「ん」。割と前から三重や和歌山では「みやん」が分布。「見」+否定として。最近,大阪でも若者が「みやん」と言い始めた。その後,「こやん」という語彙もできてきた。「来」+否定。しかし,これが時空間的にどう拡散したのかはわかっていなかった。今回これを調べた。データとして約2万のtweets(2012年1月から2015年3月)を利用。四半期別・都市別にユニークユーザ数をカウント。15-29歳の人の割合,人口密度,面積によって説明するモデル。半分以上ゼロなので,zero-inflated modelに。Spatial autocorrelationも補正(地理加重回帰?)。Moran's Iをチェックすると,2012年Q3以降は有意な正の空間自己相関が見られた。INLAパッケージを使ってzero-inflaed spatio-temporal Poisson regression。残差を使ってもう一度Moran's Iを見ると値は小さくなっていたので,空間要素を入れることで説明力が上がったと考えられる。時間要素も影響するが,空間要素の方が大きいという分析結果も出た。面白い。「こやん」はどこから? これって文化伝播の話だから,人口学会で池さんが発表していたような,反応拡散方程式を使った進行波を当てはめられるのでは? とふと思った。「こやん」が関西圏の北の方には広がらなかったのは,「へん」「ん」に社会的アイデンティティがあったからかも? という推論。谷村さんから,空間ウェイトマトリックスは隣接しているかどうか/距離で良いのか? 人の交流なのでtrip調査(パーソントリップ調査のこと?)のようなデータで移動時間を使ってマトリックスを作れば,より現実的になるのでは? と提案があったのは,もっともだと思った。
8番目はRによる探索的財務データ解析という演題。2014年に発表した論文のデータベースを更新したので,同じ方法論で分析し直して知見が再確認できるのかを試した。KGUSBADESという関西学院大学商学部で開発・運用してきた財務データ抽出システム。いくつか課題があったのでSKWADという名前でリニューアルした。来年度からそれで運用する予定。今回は,日経NEEDSからデータを抽出。dbplyrパッケージで。本来はパネルデータだが時間を止めた分析のみ示す。Time-seriesのプロットと3月期のヒストグラム。線形回帰では決定係数は70%くらいだが残差プロットがダメだったので対数変換して分析すると決定係数は90%くらいまで上がる。残差プロットをすると誤差の正規性がなく外れ値がある。4つの巨大資産をもつ会社を除去して回帰分析したらフィットは改善し,残差も正規分布に近くなった(不十分だが)。Log-skew-normal分布を仮定するともっと良く合う。t分布でもfitは良い。AICを使うとlog-skew-normalが最もよくfitしていた。質疑でbox-cox変換やM-推定が良いのでは? というコメントがあった。
9番目(今日の最後の発表者)は,樋口さんが,Rによるデータウェアハウスの利用というタイトルで。TargetMineはデータウェアハウスの1つ。エンリッチメント解析などができる。メタボローム解析というかゲノム疫学というか,そういう分野なので,理解に多くの前提知識を要し,難しい。NCBI GEOにSARS-CoV-2の遺伝子発現データが掲載された(7月22日)。RandomForest (RF)には選抜解の不安定性という問題(multiplicity)がつきまとう。RFでの変数選抜・教師無し学習(UCBの~breimanのページ参照)→エンリッチメント解析を反復という方法論を試した。BioconductorにあるInterMineRというパッケージの使い方など。それなりにうまくいったという話だが,難しくて良くわからなかった(なので質問もできなかった)。
というわけで,今年のR研究集会もオンラインだが無事終了。ありがとうございました。
R-4.1.0リリース
CHANGES IN R-develにNEW FEATURESとして"R now provides a simple native pipe syntax |> as well as a shorthand notation for creating functions, e.g. \(x) x + 1 is parsed as function(x) x + 1. The pipe implementation as a syntax transformation was motivated by suggestions from Jim Hester and Lionel Henry. These features are experimental and may change prior to release."が掲載されていた。仕様が固まった模様。4.0.4か4.1.0かわからないが,次のバージョンから導入されるか?(2020.12.11)
Rの拡張を書く(Writing R Extensions)というマニュアルがあり,パッケージやドキュメントの書き方や他言語とのAPIなどについて説明されているのだが,いつの間にかターゲットが「このマニュアルは開発中のR-4.1.0(2021年1月28日現在)のためのものです(This manual is for R, version 4.1.0 Under development (2021-01-28))」となっていた。それだけR-4.0.?からR-4.1.0への変更が大きいということだろう。(2021.1.30)
R-4.1.0が5月18日リリース予定というメールが流れた。そういえば,今年のuseR!はオンライン開催だから登録費も安いし,講義や会議の合間でも参加できそうだから参加したいなあ。登録は4月20日開始予定。(2021.4.16)
パイプ処理をネイティブ実装したR-4.1.0のソースコードが予定通りリリースされた。ちなみにコード名"Camp Pontanezen"は,相変わらずPeanutsからの引用らしい。フランスの基地で65000人の兵士がインフルエンザに罹ったって聞いて,あなたも熱あるけど……と心配されたスヌーピーが,これはインフルエンザか恋のどっちかだろうね,症状同じだから,と答える話。さらに調べると,このフランスの基地は,第一次世界大戦で米軍が引き返した場所らしく……ということは,この「インフルエンザ」はスペインかぜのことなんだな。(2021.5.18)
R-4.1.0は既にWindows版バイナリもCRANのクラウドサーバからダウンロード可能な状態になっていたので,ダウンロードはしてみた。esophデータが正しくなった(これまでesoph$ncontrolsは0であるべきところにそうでない値が入っている行がたくさんあった)というのは重要な更新ポイント。ただ,あまりにも新機能が多すぎて,メインで使う前にはいろいろチェックしなくてはいけなそう。とりあえずインストールはできたし,EZRも起動はできた。
このtweetから始まるスレッドが面白い。R-4.0.5以前はパイプ処理したい人はtidyverseというかdplyrというかHadley Wickham師の構築した世界で%>%を使ってきたわけだが,R-4.1.0で本体に|>が実装されたので,さてどうなる? という。
R-4.1.0で導入された新しいパイプ演算子|>の演算優先順位は? という質問がR-helpにあって,Duncan Murdock師による明快な回答があった。"*"より上だが":"より下とのこと。まあ,曖昧なら括弧を付ければ良いわけだが。
R-4.0.5リリース
R-4.0.4の次はパイプなどが導入されて大幅に機能追加されたR-4.1.0が来るかと思っていたが,R-4.0.4で入ってしまった2バイトコード関係のバグフィックスのためのR-4.0.5がリリースされた。(2021.3.31)
R-4.0.4リリース
Rの新バージョンはパイプや関数定義の略記などの新機軸を含む4.1.0になるかと思っていたら,その前に2月15日にR-4.0.4(コード名Lost Library Book)がリリース予定とのアナウンスがPeter DalgaardからR-develメーリングリストに流れて大騒ぎ。(2021.1.21)
R-4.0.4は予告通りリリースされたようだ。(2021.2.15)
[Rd]メーリングリストに,Hiroaki Yutaniさんからのバグレポ。R-4.0.4のWindows版とMacOS版でprint()が正しく漢字を表示してくれず\u****表記になってしまうけれどもmessage()とかcat()は正しく表示してくれるという。4.0.3ではそんなことは起こっていないので,4.0.4になったときのunicodeの内部テーブルの拡張(絵文字とか扱えるようになったらしい)の影響なのだろう。
R-4.0.4のprint()でマルチバイト文字がエスケープされてしまう件,通称R教授が対応するけど数日かかると返事していたが,裏RjpWikiさんの情報によると直ったとのこと。まあしかし,今さらPatched版のR-4.0.4をインストールするのもどうかと思うので,R-4.1.0リリース待ちかな。(2021.2.23)
操作変数法
操作変数法をRで実行する方法として,semパッケージでtsls()を使う方法と,AERパッケージでivreg()関数を使う方法を講義資料の7.4(pp.116-8)で説明しておいたが,これらは目的変数が正規分布に従う線型モデルでしか使えないので,ロジスティック回帰で実行する方法を探してみたら,ivtoolsパッケージのivglm()関数や,naiveregパッケージのLIVE()関数またはnaivereg()関数でできそうだった。Rのパッケージ凄いな。
『サラッとできる! フリー統計ソフトEZR(Easy R)でカンタン統計解析』
メールボックスを見たら,神田善伸先生の新刊『サラッとできる! フリー統計ソフトEZR(Easy R)でカンタン統計解析』オーム社,ISBX978-4-274-22632-8が入っていた。石田先生の弁当屋シリーズに近い雰囲気で,親しみやすく書かれていた。唯一残念だったのは,MacOSへのインストール方法が「上記のホームページをご覧ください」になっていた点。EZRを大学院生向けの講義で使っていて,最大の難関は,院生のPCがMacである場合で,自治医大のサイトの説明通りにやっても上手く行かないという学生が毎年続出するのだ。MacOSのバージョンやX11のバージョンなどいろいろ面倒な条件があるので,すべてに対応してわかりやすい解説をつけるというのは至難の業だが,それがあったら入門書としては完璧に近かったのに,と思う。しかし,分布やクロス集計の話から始まって最後はロジスティック回帰や生存時間解析までカバーしていて,マンガやエピソードも無理がなく,全体としては良い本だと思ったので,学生に薦めてみよう。ご恵贈ありがとうございました>神田先生。
R-4.0.3リリース
R Core TeamのPeter Dalgaard先生から,R-4.0.3が10月10日リリース予定というtweetがあった。コードネームは"Bunny-Wunnies Freak Out"と,PeanutsのBunny Wunnyシリーズの,1972年10月23日掲載のものから取られたようだ。"THE SIX BUNNY-WUNNIES FREAK OUT"という本(これに限らず,BUNNY WUNNYシリーズは架空の本だが,スヌーピーが大好きなシリーズという設定になっているらしい)が学校の図書館で閲覧禁止になって困惑しているというストーリー。R-4.0.3にこのコードネームを付けた寓意は何だろう?
R-4.0.3が予定通りリリースされた。
R-4.0.3 ("Bunny-Wunnies Freak Out"),クラウドサーバからWindowsバイナリがダウンロードできた。マイナーアップデートなので機能追加はほとんどないが,結構いろいろなバグフィックスがされているようだ。
RStudioでの日本語コードと開発コード名
1つメモしておくと,RStudioはutf-8しか許さないのかと思っていたが,Tools>Project Options>Code Editing>Text Encodingでマルチバイトコードを変えられる。主にRtermでバッチ処理するため,日本語文字コードをShift-JISにしているフォルダのプロジェクトで,ここをCP932に指定したら,RStudioとRtermで文字コードが違う問題が解決した。
ふと思い立ってRStudioのリリースノートのページを見て気づいたが,バージョン1.2の2019年11月1日版から花の名前のコードネームがついているんだな。最新版はApricot Nasturtiumなので,杏色のキンレンカと思われる(WikipediaによるとNasturtiumは本来オランダガラシを指す名前だったそうだが,杏色の花ならばキンレンカで良いのだろう)。R本体のコードネームがPeanuts!由来なのも洒落ているが,RStudioの花の名前も粋だな。
R Studioの1.4(リリースノートによると,1.4.1103,2021年1月14日付け,コード名はWax Begonia)がリリースされたのでダウンロードしてみた。書類ができたらインストールする。
いつの間にかRStudioが更新されていた。Release Noteによると、コード名が"Ghost Orchid"(ナショジオによると幽霊ランという、きわめて見つけにくい植物らしい)なのか"Prairie Trillium"(エンレイソウ)なのか良くわからないが、ともかく2022.02.0+443版は(バージョン番号がv1.4.1717-3までとは違って、たぶん西暦年.月.月内更新番号+マイナー更新番号と思われる命名規則になった)、UCRTに対応し、グラフィックエンジンも15に対応したので、UTF-8にネイティブ対応しているWindows版R-4.2.0にも対応したとのこと。早速インストールした。(2022.2.18)
useR!2020
今年のuseR!はセントルイスでは実施せず,ヨーロッパハブと合同でオンライン開催すると発表されていて,詳細プログラムも出ていて,基調講演などはZoomで参加でき(詳細プログラムからリンクされている),一般発表はキュレーションされた後YouTubeで公開されるようだ(Tweet)。
効果量
大学院生からメールでKruskal-Wallis検定の効果量についての問い合わせがあって調べてみたところ,rcompanionとstatsExpressionsという凄いパッケージ(それらのターゲットとなるhtmlテキストとpdf版テキストと,その元となる教科書のpdf版)があって,epsilonSquared()関数を使う方法がわかった。他には,rstatixパッケージのkruskal_effsize()関数を使うこともできるようだ。chickwtsデータフレームで,使い方の例を挙げておこう。
boxplot(weight ~ feed, data=chickwts)
kruskal.test(weight ~ feed, data=chickwts)
library(rcompanion)
epsilonSquared(chickwts$weight, chickwts$feed, ci=TRUE)
とすると,ε2の推定値と,ブートストラップ法で求めた95%信頼区間まで出してくれて,0.533 [95%CI 0.391, 0.713]とわかる。statsExpressionsのリンク先によると,ε2が0.26以上なら効果量大とのこと。
昨日のε2の件を含め,ANOVAとKruskal-Wallisの検定の効果量について説明を追加し,講義テキストを更新した。
Harrer, M., Cuijpers, P., Furukawa, T.A, & Ebert, D. D. (2019). Doing Meta-Analysis in R: A Hands-on Guide. https://bookdown.org/MathiasHarrer/Doing_Meta_Analysis_in_R/.を見つけた。Rのリソースは日々充実しているなあ。多重比較の効果量についても院生から質問があって,いろいろ調べていたら,Research Gateで2014年に行われていた議論でHedge's gが良いという意見もあったが,2016年に出た論文が一般化η2について検討していて,デザインをちゃんと考えた上で使うべきと述べていた。ぼくは効果量より信頼区間派なので,多重比較なら同時信頼区間を出す方が良いと思うが。
反復測定分散分析の効果量計算方法について質問メールがあり,調べてみたところ,まず,RcmdrPlugin.aRnovaパッケージのrepMeasAnova()関数でできそうだとわかった。これはRcmdrのプラグインなので,インストールするとRmcdrのメニュー(EZRではメニュー右端の標準メニュー)の,統計量>平均>反復測定分散分析から計算できそうに思われた。ところが,オプション指定の方法がわからない。2018年3月以降チュートリアル文書なども途中で開発が止まっていて(GitHubページもバージョン0.0.5以降更新がない),使い方を書いたものがどこにも見つからないのだった。そこで別の方法を再検索したところ,井関龍太さんが開発されているanovakunというRのコードに辿り着いた。反復測定デザインにおける効果量を参照すると,計算式自体についても議論があるようだが,とりあえず一般化イータ二乗を出せば良かろうと思われた。そこで,自分の講義テキストの158ページに,EZRでの反復測定分散分析の実行例を示してある例で試してみた。anovakunはパッケージになっていないので,まず,このページに書かれているように,テキスト形式のコードをダウンロードしてsource()で読み込むか,あるいはエディタで開いて全部選んでコピーしてからRコンソールに貼り付けるなどして,anovakun()という関数が使える状態にする必要がある。その上で以下のコードを実行する。
> x <- read.delim("https://minato.sip21c.org/ogtt02.txt")
> anovakun(x[, -2], "AsB", 2, 8, geta=T)
ここで与えるデータをx[, -2]としているのは,2列目のIDという変数が余計なので削除するためで,"AsB"というのは効果を見たいグループAの変数が1列目にあり,それ以降の列がすべて時点ごとの測定値である場合に指定する文字列で,2はグループAには2水準(ControlとObesity)あることを意味していて,8は時点が8時点あることを意味している。geta=Tが一般化イータ二乗を計算させるためのオプションで,TはTRUEと打つ方が良いと思うが,井関さんのサイトではTで書かれている。これを実行すると長い出力が得られるのだが,わりと上の方に下表が出てくる。Aの行がグループの主効果,Bの行が時点の主効果,A x Bの行がグループと時点の交互作用効果を示していて,右端のG.eta^2が一般化イータ二乗である。
----------------------------------------------------------------
Source SS df MS F-ratio p-value G.eta^2
----------------------------------------------------------------
A 15.3392 1 15.3392 6.6592 0.0148 * 0.1229
s x A 71.4070 31 2.3035
----------------------------------------------------------------
B 39.2486 7 5.6069 31.9359 0.0000 *** 0.2638
A x B 7.7287 7 1.1041 6.2887 0.0000 *** 0.0659
s x A x B 38.0984 217 0.1756
----------------------------------------------------------------
Total 175.2562 263 0.6664
R-4.0.2リリース
R-4.0.1のメモリ解放に関わる問題の話だが,Core TeamのPeter Dalgaard教授から6月22日にR-4.0.2をリリースするというアナウンスがあった。少なくともWindows環境ではR-4.0.1はインストールしないのが吉。(2020.6.10)
R-4.0.2(コード名Taking Off Again「再び飛び立ってゆく」,という虫の行動を観察しているスヌーピーの哲学的な独白だが,今回R-4.0.1で手痛いバグに悩まされたことから立ち直ろうという寓意があるのだろうか?)が予定通りリリースされたというアナウンスがあった。
帰宅して晩飯を適当に作って食べ,cranを見てみたら,既にR-4.0.2がWindowsバイナリもダウンロードできる状態になっていたので,インストールしてみた。パッケージはR本体の下ではなく,R_libs環境変数を設定してe:/Rlibsに入れてあるので,インストールし直す必要は無く,update.packages(checkBuilt=TRUE, ask=FALSE)で更新したら済んだようだ。R-4.0.2では,EZR/Rcmdrのtck/tk絡みの問題はほぼ解消した感じで,EZRでデータ編集ができない問題は直っていた。アクティブデータセットの切り替えが,リストの一番下のデータセットを選んだときだけ,操作対象は切り替わるのに表示が変わらないという問題が起こったが,もう一度試してみたら再現しなかったので,バグレポートもできず困った。
Microsoft R Openが2020年9月に4.0.2をリリースしていたのを見逃していた。ここからダウンロードできる。ただ,R本家が近々4.1.0をリリースしそうなので,このタイミングでMicrosoft R Openの4.0.2を入れる気はしないが。(2021.1.5)
『Rで学ぶ人口分析』→『Rによる人口分析入門』
出版社から『Rで学ぶ人口分析』の再校が届いた。家にいて良かった。なお,数日前にサポートページのコードはR-4.0.0での動作確認が済んでいて,章ごとに整理して掲載するという作業が済んでいる。
出版社から連絡があり,本のタイトルは『Rによる人口分析入門』となり夏の発売とのこと。初版1000部だそうだ。自分としては「Rによる」より「Rで学ぶ」の方が好みなのだが,出版社の判断なので仕方がない。いずれにせよ,校正は急がないと。(2020.6.8)
朝倉書店から8月刊行予定と書影付きでtweetされ,価格や目次も出たので,これは情報オープンにして良いということだろう(サポートページもリンクしてくださっているので,タイトル,出版社,ISBN,価格の情報を載せねば)。再校締め切りは今週末なのだが,今日中に終わらせよう。
そろそろ店頭に並ぶと思われる『Rによる人口分析入門』,川端君が感想をtweetしてくれた(その後も前書きについて,国勢調査と人口動態統計について,死亡と出生について,「お絵かき」もできる件,といろいろ書いてくれてありがたい)。(2020.8.27)
もう9月1日か。『Rによる人口分析入門』発行日だが,多くの人が読んでくれたら良いなあと思う。(2020.9.1)
傾向スコア
午後は4限に保健統計学特講という,保健師コースの大学院生5人だけを対象とした講義の分担2回のうちの1回目。傾向スコアの話にも軽く触れるが,時間がないので,東北大学の宮田先生の講演資料とか,東京大学の康永先生のm3記事とか,Rのtwangパッケージのmnps()関数を使って傾向スコアマッチングをする方法のチュートリアルとかiptw()関数を使って反復処理のために傾向スコアを使う方法のチュートリアル辺りを紹介して,後は読んでおいて貰うか。
KRSKさんがtweetで紹介されていたJournal of Epidemiologyの傾向スコアのチュートリアル論文は素晴らしいと思う。全文公開なのも素晴らしい。Supplementary Materialも見るべき。
『効果検証入門:正しい比較のための因果推論/計量経済学の基礎』
安井翔太(著)株式会社ホクソエム(監修)『効果検証入門:正しい比較のための因果推論/計量経済学の基礎』技術評論社,ISBX978-4-297-11117-5は,1月末に入手したのだが,ずっと読む暇が無かった。けれども,傾向スコアとかDIDとかRDDを丁寧に例を使って説明してくれているし,とくにDIDについて,疫学では有名なSnowの自然実験に出てくる2つの水道会社(S&V社とL社)別のコレラ死者について,Snowが使ったどちらの会社からの給水もある地区での1854年のコレラ死亡率の会社間比較ではなく,L社もテムズ川下流から採水していた1849年からの変化を2社間で比較するためにDIDを使って分析する方法を示してくれているのが面白い。
rdhsパッケージ
RでDemographic and Health Surveysのデータを利用するためのパッケージrdhsというものがあるのを見つけた。もちろんDHSへのユーザ登録を予め済ませておく必要があるが,DHSでダウンロードできるデータ形式はテキストファイルかSTATA形式かSAS形式かSPSS形式なので,このパッケージを使う方が便利かもしれない。ともあれ,かつて登録したアカウントがまだ生きていたので,インドネシアの2017年のDHSデータをダウンロードした。とりあえず火曜午後の保健統計学で示す例として使ってみよう。
R-4.0.1リリース
R-develメーリングリストに,R-4.0.1が6月上旬,たぶん6/6リリースというメールが流れた。
いつの間にかRStudioのフリー版デスクトップの1.3.959がリリースされていたので,ダウンロードしてインストールした。
R-4.0.0(コード名Arbor Day)が4月24日にリリースされてから,そんなに日は経っていないが,R-4.0.1(コード名See Things Now―出典はこれ?)が6月6日リリース予定というアナウンスが出た。
R-4.0.1(コードネーム:(眼鏡を掛けたおかげで)今はモノが見える[See Things Now] ― 出典)がリリースされた。(2020.6.6)
R-develメーリングリストで,RcmdrのメインテナでもあるJohn Foxから,R-4.0.1はtcltkパッケージを使うと落ちるという報告があり,Duncan MurdochやPeter Dalgaardらコアチームのメンバーを含めたメールが飛び交っている。メモリ解放関係の問題っぽい。RcmdrやEZRはR-4.0.0になってからうまく動作しないことが何度かあったが,R自体が落ちるとは。RcmdrやEZRを主に使う人は,R-3.6.3のままにしておくべきかもしれない。John Foxはこの問題が解決するまでR-4.0.1のWindowsバイナリは公開を取りやめておく方が良いのでは? とまで言っている。(2020.6.8)
Core TeamのPeter Dalgaard教授からのメールで,WindowsのR-4.0.1でtcltkを使いたい人には,(1) R-4.0.1-patchedを使うようアナウンスする,(2) Windows版のメインテナがVendor-patch-releaseとしてバイナリを再アップロードする,(3) 最短でR-4.0.2をリリースする(現実的には6月22日)のどれかしかないとあった。うーむ。(2020.6.8)
Lotka-Volterra移植
数理生物学会のニューズレターが届いていて,Python実行環境としてAnacondaとJupyterのインストール方法とLotka-Volterraの被食捕食関係のモデルの数値計算コードの実行の仕方の解説記事が載っていたので,やってみたくなった。が,もちろんRでも簡単にできるので,なるべく元のPythonコードから変えずに移植してみた。いろいろ簡略化できるところはあるが,まあ,とりあえず。
R-4.0.0リリース
明日公開されるはずのR-4.0.0の変更点がメチャクチャ多い。すぐ導入すると問題起こるかもなあ。(2020.4.23)
R-4.0.0リリースのアナウンスが来た。既にソースコードはCRANからダウンロードできる。
R-4.0.0では3.6.3までで使っていたパッケージを全部再インストールしなくてはいけないのだが,それを自動でやってくれるスクリプトを作って公開している方がいた。このtweetからリンクされているGithubのRコード。
anovakunがR-3.6.3までのコードではR-4.0.0で正しく動かず,R-4.0.0用に修正したらR-3.6.3以下では正しく動かないと書かれていたので,「大変とは思いますが,可能ならR.Version()$major>=4で条件分岐して旧コードと新コードを1つにまとめれば,後方互換性が保てるのではないかと思います」と引用tweetした(注:括弧を付け忘れたので返信tweetで修正したが)。ソフトウェアは,パッケージも含めて,できるだけ後方互換性は保っておく方が良いと思う。survivalパッケージのsurvfit()が1群の場合に"~1"を付けないとエラーになるという仕様変更がされたときは困った。UIが変わらなければ中身は後方互換でなくても問題ないのかもしれないが。あるいは,kohskeさんの返信tweetのように,options(stringsAsFactors = TRUE)だけで対処できるなら,もちろんその方が良いと思うが。しかし自分のパッケージも動作確認必要だろうなあ。人柱になるしかないか。
というわけで,R-4.0.0をインストールした。インストールすべきパッケージは全てinstall.packages()に打ってRコードとしてまとめてあるので,それを実行したのだが,それだけだとダメだった(実際にlibrary(Rcmdr)しようとすると,足りないパッケージがあると表示されて動かない)。そこで,update.packages(checkBuilt=TRUE, ask=FALSE)を実行したら,今度は全部更新されたようで,無事にEZRも動作したし,RStudioで前回のDemographyの講義で使ったコードを実行することもできた。mapdataと組み合わせて地図上に人口ピラミッドを描くコードもうまく動いたのでpyramidは大丈夫そうだが,fmsbのすべての関数のチェックはすぐにはできないなあ。
RStudioでfmsbパッケージのprojectを開いてCheck packageをすると最後にRtoolsがないというエラーが。rtools40(R-4.0.0以降の開発のためのRtools)ではパスをe:/rtools40/usr/bin(R-3.6.3までに対応していたRtools-3.5まではe:/Rtools/bin)としなくてはいけないのだった。binがusrの下に入ったことに気づかなかった。Windows環境での開発者は要注意。なおEドライブなのはCドライブが小さすぎるからで,たぶん普通Cドライブにおくだろう。Check自体はノーエラーで通った。radarchart()など,わりと複雑なことをしている関数のexample()も問題なく表示された。でもやっぱり,testthatパッケージを使っておくべきなんだろうなあ。Hadley Wickham師による"R packages"の第10章を読みながらやってみるか。
testthatの使い方は,パッケージフォルダの中にtestsというフォルダを作り,その中にtestthatというフォルダを作り,さらにその中にファイル名がtestで始まるRコード(例えばtestthat.R)を作る。testthat.Rの中に書くファイルの文法はそんなに複雑ではないが,expect_equal()で実数と等しいかどうかを判定するときは,1e-8の桁まで合わないとテストを通らないのが面倒だな。差を取ってround()して比較するか?
R-4.0.0にしてしまったので,明日の人口学講義で使うRコードのチェックをした。options(stringsAsFactors=FALSE)の動作がデフォルトになってしまった(理由はKurt Hornikさんのブログ記事に書かれていた)ので,横軸に年齢階級をとったplot()が従来のコードでは正常動作しなくなってしまった。先頭でoptions(stringsAsFactors=TRUE)をするのが一番楽だが,それももうすぐ動作しなくなると書かれていたので避けたい。しかし読み込んだ後でas.factor()で型変換するのも鬱陶しい。一番楽な解決策は,文字列を読んだときに自動的にファクター型にさせたいread.delim()の中にstringsAsFactors=TRUEをオプションとして書くことであった。
順序カテゴリ変数の分布の差の尤度比検定
順序カテゴリ変数の分布に2群間で差があるかどうかを解析したいときにどうしたら良いか,数日前に院生から相談を受けて返事した内容,ここにもメモしておく。よくされているのは,順序カテゴリだが元々連続量だったものを区間でカテゴライズしたものであるならば,区間の中央値で代表させ,2群間で平均値の比較をしてしまうとか,順序のままWilcoxonの順位和検定をするとか,順序の情報を無視してカイ二乗検定するとかだが,もっと良い方法として,このページに説明されているように,グループで順序を説明するという順序ロジスティック回帰の結果と,定数で順序を説明するという順序ロジスティック回帰の結果の尤度比を取る方法が考えられる。例えば,
set.seed(123)
y1 <- c(sample(1:3, 20, rep=TRUE), sample(4:6, 30, rep=TRUE))
y2 <- c(sample(1:3, 30, rep=TRUE), sample(4:6, 20, rep=TRUE))
y <- as.ordered(c(y1, y2))
x <- as.factor(c(rep("A", 50), rep("B", 50)))
table(y, x)
library(MASS) # polr()関数を使うので
anova(polr(y ~ x), polr(y ~1)) # 尤度比検定
chisq.test(table(y, x)) # カイ二乗検定でもやってみる
を実行すると,尤度比検定では5%水準で有意差ありだが,順序を無視したカイ二乗検定では有意差なしとなる。
COVID-19関連
COVID-19だけの話ではないが,Prof. Marc Lipsitchのグループが書いた論文,McGough SF et al. "Nowcasting by Bayesian Smoothing: A flexible, generalizable model for real-time epidemic tracking."(2019年6月7日)が,PLOS Computational Biologyに載る予定とのこと。そこで使った計算コードがNobBSというパッケージとして整備され,2020年3月3日付けでCRANに載った。(2020.3.7)
R-develメーリングリストで,covid-19についてデータをダウンロードして分析できるようなサイトを教えて欲しいという投稿があり,これ(githubページ)とかこれ(CSV形式のデータそのもの)とか,これ(これもgithubページ)といった情報が返ってきている。(追記)1つ追加された。(2020.5.5)
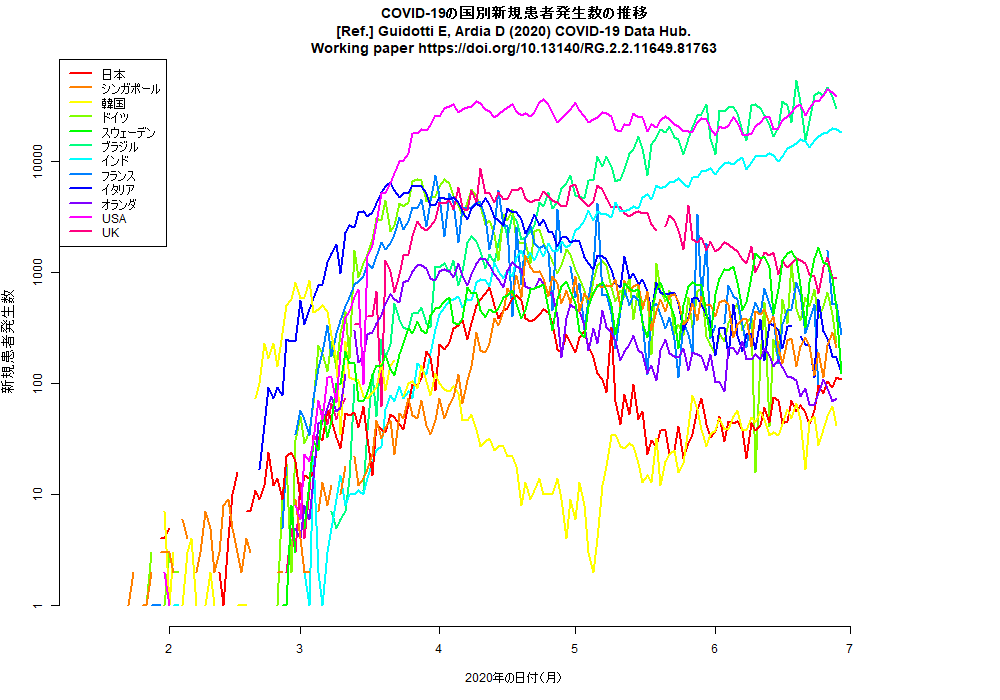
5月5日にリンクしたが,R関係のリソースは使いやすくて,国際比較のデータを読んで片対数グラフを描くコードくらいなら,このように(リンク先は先に示したコードの実行結果PNG画像)簡単にできる。ちなみに,このコードで工夫したのは,ローカルにデータファイルがあればそれを読み,無ければダウンロードしてから読んで描画するようにした点。(2020.5.9)
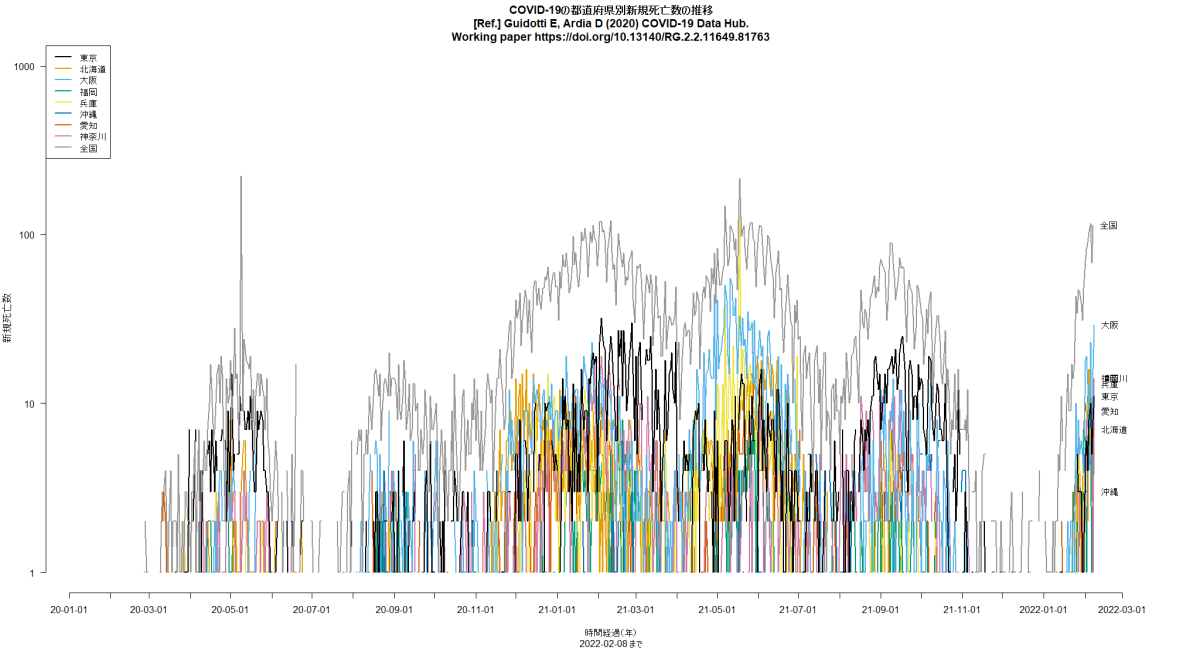
一昨日は確定患者数について国際比較するコードを書いたが,今朝,ふと思い立って死者数について書いてみた。deaths.Rとworlddeathssemilog.pngをリンクしておく。Rコードにコメントしてあるが,データは,COVID19パッケージのgithubページからリンクされているcsvファイルを使った( Guidotti E, Ardia D (2020) COVID-19 Data Hub. Working paper https://doi.org/10.13140/RG.2.2.11649.81763)。ちなみに,使ったcsvファイル(data-1.csv)はレベル1,つまり国レベルの情報で,累積確定患者数(confirmed),累積死者数(deaths)だけではなく,累積検査数(tests),累積治癒数(recovered),各日における入院患者数(hosp),各日におけるICU入院患者数(icu),各日における侵襲のある人工呼吸器を必要とする患者数(vent),学校閉鎖状況(0: 対策無し,1: 閉鎖推奨,2: 部分的閉鎖要請,3: 全面的閉鎖要請)(school_closing),職場閉鎖状況(0: 対策無し,1: 閉鎖または在宅勤務推奨,2: 部署あるいは職種により閉鎖要請,3: 社会維持に必須な職場を除き閉鎖または在宅勤務要請)(workplace_closing),イベントキャンセル(0: 対策無し,1: イベントキャンセル推奨,2: イベントキャンセル要請)(cancel_events),集会制限(0: 制限なし,1: 1000人以上のマスギャザリング制限,2: 100人以上の集会制限,3: 10人以上の集会制限,4: 10人未満でも集会制限)(gatherings_restrictions),交通停止(0: 対策無し,1: 交通停止あるいは減便推奨,2: 交通停止あるいは大部分の市民について利用禁止要請)(transport_closing),外出制限(0: 対策無し,1: 外出非推奨,2: エクササイズや生活必需品購入のためを除き在宅要請,3: 2-3日に1度一人だけという最小限の例外を除き在宅要請)(stay_home_restrictions),国内移動制限(0: 対策無し,1: 閉鎖推奨,2: 閉鎖要請)(internal_movement_restrictions),国際移動制限(0: 対策無し,1: スクリーニング,2: ハイリスク地域からの到着者について検疫,3: ハイリスク地域について出入国禁止,4: 全面的国境閉鎖)(international_movement_restrictions),情報キャンペーン(0: なし,1: 公的注意喚起,2: 複数セクタが協力した公的情報キャンペーン)(information_campaigns),検査政策(0: なし,1: 症状がありかつ特定基準を満たす人だけ検査,2: 症状があれば誰でも検査,3: 無症状でも誰でも検査)(testing_policy),接触者追跡(0: なし,1: 限定的接触者追跡,2: 全ての症例について包括的な接触者追跡)や,人口の情報(popが総人口,pop_femaleが女性人口割合,pop_14が年少人口割合,pop_15_64が生産年齢人口割合,pop_65が老年人口割合)なども含まれている。しかし,social distancingの程度とか三密予防をしているかといった情報は含まれていない。レベル1の他にレベル2のstateレベルのファイルとレベル3の市町村レベルのファイルがあり,COVID19パッケージを使うと,例えば,covid19(c("ITA","USA"), level = 3)でイタリアとUSAの市町村別データが得られるなど,どのレベルの情報でも読めると書かれている。(2020.5.11)
https://www.datacat.cc/covid/で作ったグラフはメディアへの掲載が許されないらしく,Rでグラフを作った。記事にURLが載っているコードだと英語表示なのと,いつの間にかデータファイルからcountryという変数が消えてしまったのでadministrative_area_level_1をcountryに付値し,メッセージも日本語化したコードを載せておく(Shift-JIS版とUTF-8版)。国名を変えたり増減させるだけで他の国も灰色でない表示にできるはずなので,例えばアイスランドよりブラジルを表示したかったら"Iceland"を"Brazil"に,"アイスランド"を"ブラジル"に変えるだけで,自動的にアイスランドの線が灰色になり,ブラジルの線にマゼンタ色が付く。(2020.5.17)
cranにR0というパッケージがある。いくつかの方法がサポートされていて,講義には便利かもしれない。(2020.5.22)
奥村先生のtweetで知ったが,Stutt ROJH et al. "A modelling framework to assess the likely effectiveness of facemasks in combination with ‘lock-down’ in managing the COVID-19 pandemic." Proc. Royal Soc. A(2020年6月10日)は,マスク着用の効果を評価しているモデルで,症状がなくても一般公衆が常にマスクをしていれば実効再生産数を1未満にできて感染拡大を緩和できるとabstractに書かれている。ざっと目を通した感じでは,マスク着用するかどうかは日々刻々と変わると思うので,シミュレーションするなら個人ベースで,その振り分けを二項乱数でやった方が良いと思うし(ぼくが長袖長ズボンの着用によるマラリア防御モデルでやったように),デフォルト値がmF=1,mA=0.5,mS=0.5,mD=0.5というのは非現実的で,ウイルスが付着したものを触った手で顔を触るのを防ぐわけだから,mF=0.1とかmF=0がデフォルトであるべきだし,普通の布マスクやサージカルマスクならば,着用している人の唾液飛沫が出て行くのを防ぐ効果の方が飛沫やマイクロ飛沫が入ってくるのを防ぐ効果よりずっと大きいはずだから,mDは0.5より大きく,mAやmSは0.5よりずっと小さくなると考えるのが自然ではないだろうか。コードが公開されている(MITライセンス,いくつかの依存パッケージはあるが,ピュアなRのコード2つからなる)ので,後で動かしてみよう。
ScienceのWalker PGT et al. "The impact of COVID-19 and strategies for mitigation and suppression in low- and middle-income countries" DOI: 10.1126/science.abc0035(2020年6月12日)は,たぶんインペリグループ第12報が低中所得国にフォーカスを絞って改稿してScienceにアクセプトされたものだろう。Supplemental Materialによると,モデルはGithubでRパッケージとして提供されていて,この論文の解析コードも,Githubで公開しているとのこと。
12日に触れたマスクのシミュレーションのコードをノートPCで実行させてみたが,遅くて話にならない(どれくらい話にならないかというと,100万回のループのうち1000回で丸一日掛かったので,たぶん1000年日掛かる。CPUはCore i7なんだが)。高速なデスクトップが必要か? あるいはRcppを使って書き換えるとかで高速化するか? 3.5.3以降更新されていないのだが,Microsoft R Openにしてみるとか?
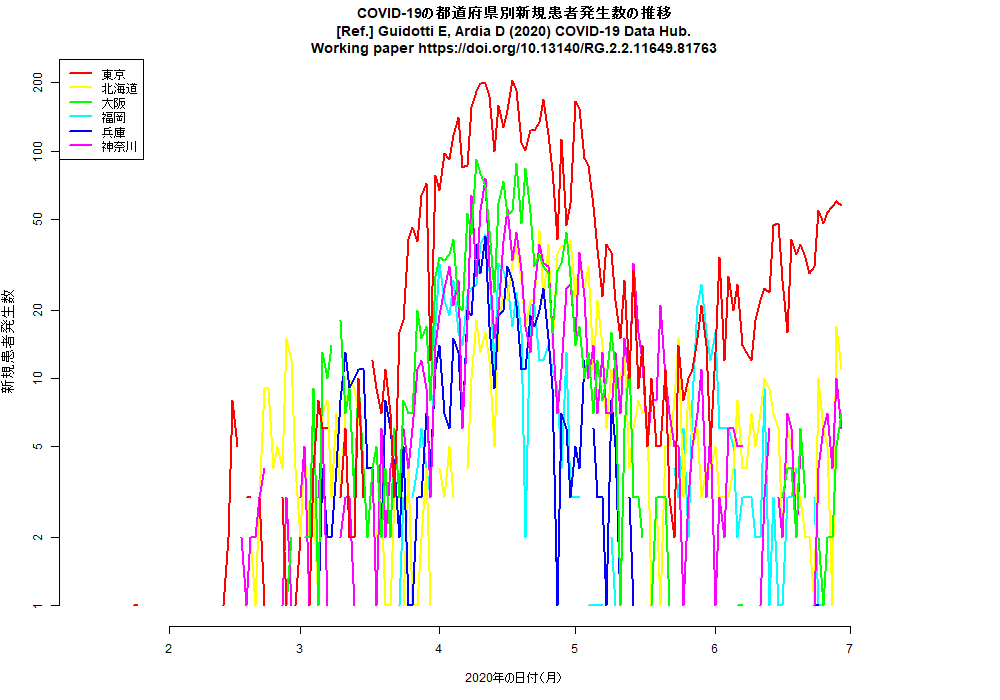
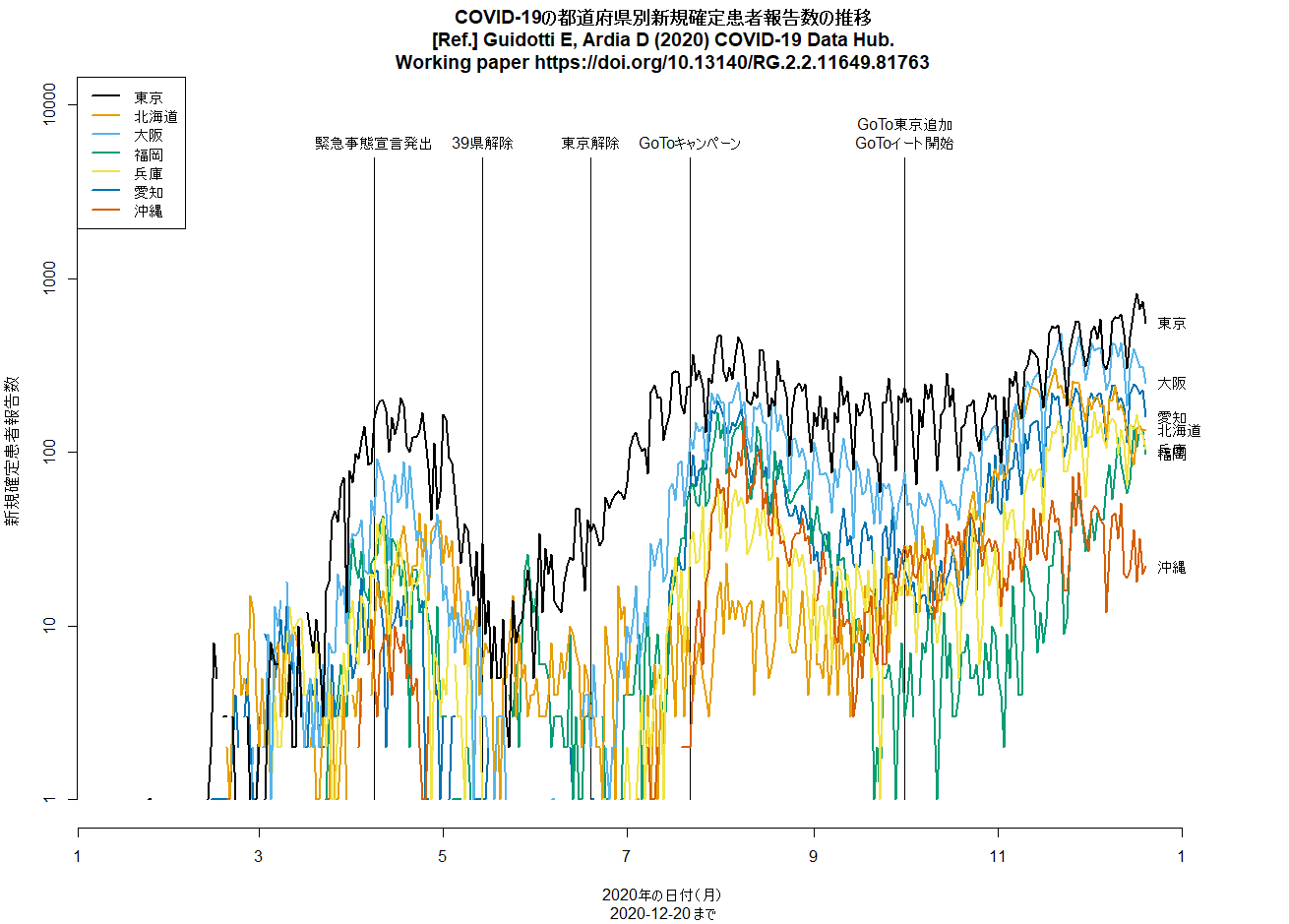
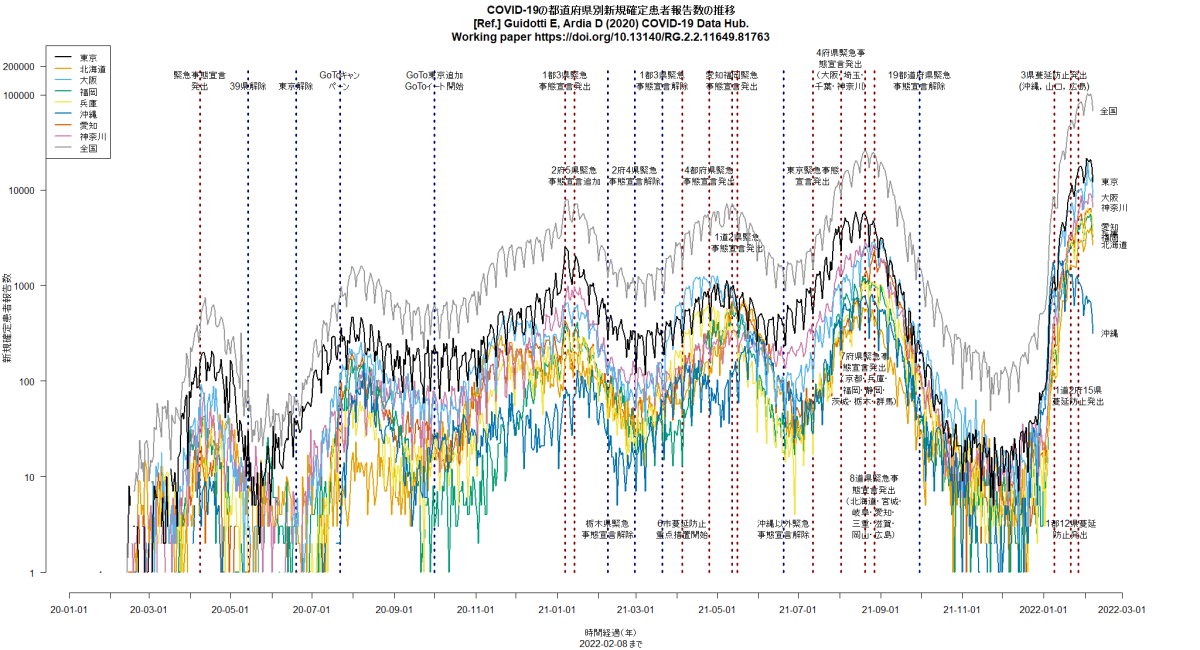
RのCOVID19パッケージを使って,日付別新規患者発生数をいくつかの国についてと都道府県について描かせてみると(コード),下図が描かれる。明らかにUSAも東京も増加傾向にある。(2020.6.28)
このtweetで紹介されている論文はプレプリント(2020年7月3日)だが,その中で計算に使われているRのEpiEstimというパッケージ(vignette)は,既にいくつかの査読を通った論文でも使われている。例えば,Ruiz-Patiño A et al. "Mortality and Advanced Support Requirement for Patients With Cancer With COVID-19: A Mathematical Dynamic Model for Latin America"(JCO Global Oncology,2020年5月29日)やNajafi F et al. "Serial interval and time-varying reproduction number estimation for COVID-19 in western Iran"(New microbes and new infections,2020年6月14日)でも使われていて,MCMCを使った再生産数リアルタイム推定の常套手段になりつつあると言って良いかもしれない。暇を見つけて試してみたい。最初に触れたプレプリント論文の著者たちは,EpiEstimを使ってCOVID-19の再生産数推定を実装するためのパッケージとして,EpiNow,EpiSoon,NCoVUtilsという3つを開発しているようだ。
全国で過去最多の新規感染確定報告というニュース。とくに大阪と福岡と愛知の増え方が目立つ。数日前までの増え方からしたら当然だよなあと思って,再度Rコードを走らせて作図してみたら,状況がわかりやすかったので,このコメント見逃していましたが,この時点で東京よりも大阪や福岡の方が片対数グラフの傾きが急だったので潜在的に危険だと思っていました。さっき神奈川の代わりに愛知を入れて再作図してみたら,やはりそうかという印象です。とtweetしてから眠った。(2020.7.29)
日本の東京や大阪などのCOVID-19の新規感染者数推移を縦軸を対数軸にして描画するRコードは,libusejapan.Rとしてダウンロードできる。COVID19パッケージを使って,実行する度にデータをダウンロードし,実行日の前日までをプロットするようになっている。(2020.11.24)
昨日のコードは日本の都道府県版だったが,COVID19パッケージを使うと世界の国々についての新規感染者数推移も簡単に描ける。libuseworld.RをRで実行すれば,いつでも,その時点の1日か2日前までのグラフができる。(2020.11.25)
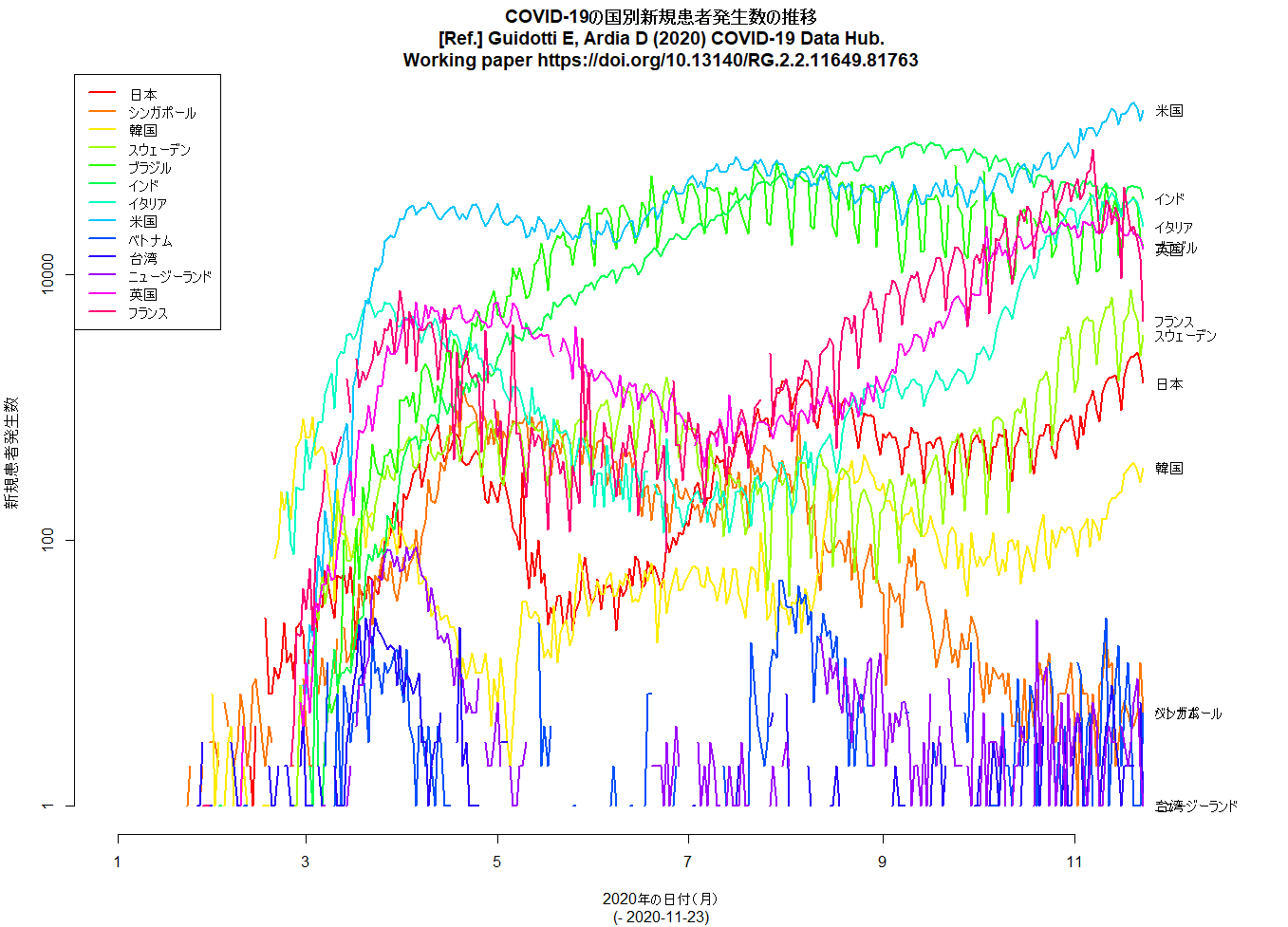
COVID19パッケージでの国別の描画には3文字の国名コードが必要なのだが,この一覧表が便利かも。
libusejapan.Rをpalette("Okabe-Ito")を使って色をsafe colorsで作図するように変えてみた。
昨日付けで,covid19jpというパッケージがcranに入っていた。GitHubに書かれている説明によると,データソースは,大変有名な東洋経済の荻原和樹さんが公開しているものとのこと。荻原さんの公開データはCSV形式なので,covid19jpパッケージは,そこにカスタマイズされたインターフェースを付け加えたということだろう。(2021.1.10)
cranのCOVID19関係のパッケージも随分増えて,ぼくはこれまでも作図のためCOVID19パッケージをよく使ってきたが,日本のデータに特化した上述covid19jpパッケージやブラジルのマイクロデータを提供しているCOVIDIBGEパッケージもできてきたし,移動の傾向をさまざまなソースから引っ張ってくるcovid19mobilityパッケージ(紹介記事)もあるし,モデルについてもSPARSMODrパッケージが入院率が変化したときの感染動態の理解に使えることを示した文書が発表されたりしているし,7月にも触れたEpiEstimパッケージを使った論文はますます増えて,Cheng Q et al. "Heterogeneity and effectiveness analysis of COVID-19 prevention and control in major cities in China through time-varying reproduction number estimation"(Scientific Reports,2020年12月15日)でも使われている。(2021.1.10)
昨年4月,Gigazineで紹介されていたVollmer教授らの年齢調整IFRで死亡数を割って求めた感染者数と確定感染者数から検出割合を計算する方法について簡単に説明したが,最近までのデータについて,この方法を適用してみると(Rコード),下図のように,検出率が異様に高くなってしまう。こんなに捕捉されているとは考えにくいので,可能性としては,(1)昨年4月に想定されていたよりIFRが下がっている,(2)感染確定から死亡までの平均期間が長くなっている――Prof. Vollmerの方法では2週間という仮定なので,確かに短すぎるだろう,(3)見過ごされる死亡が増えている,といったことが考えられる。(2021.1.24)
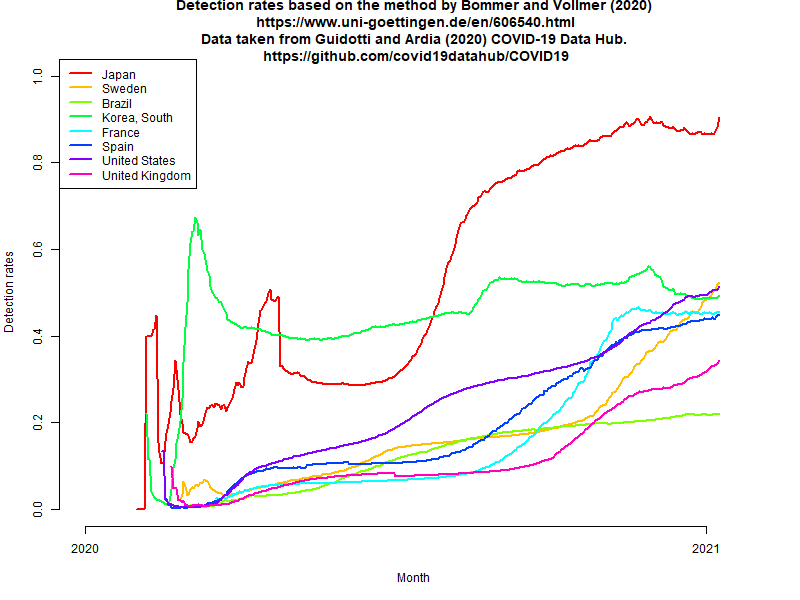
(追記)仮に上記3つの可能性がすべて否定されたら,日本の感染者はこれまで8割方捕捉されてきたことになる。それが意味するのは,(1)保健所の方々を中心とする接触者追跡により,感染者と濃厚接触して二次感染した人の(前向きの)リンクはほぼ辿れていたこと,(2)しかし時間遅れなどのため,その先の感染を防ぐことができていなかったこと,その結果が(3)年末年始の急激な感染拡大になってしまったこと,そして,最近いくつかの自治体が始めてしまったように接触者追跡を諦めたら,捕捉率(検出率)が急激に低下し,さらなる感染者増加に繋がるだろうということである。その意味で,今後数週間,この方法による検出率推定をしてみる価値はあるかもしれない。(2021.1.24)
この検討は簡単にできるので続けているが,Vollmer教授の方法で推定される捕捉率が異様に高いままなので(現在までのデータでカラーパレットをカラーユニバーサルデザインの"Okabe-Ito"に変えて描画したのが下図),おそらく実はもっと捕捉率が低い可能性(1)~(3)のどれかが現実だと思われる。(2021.2.3)
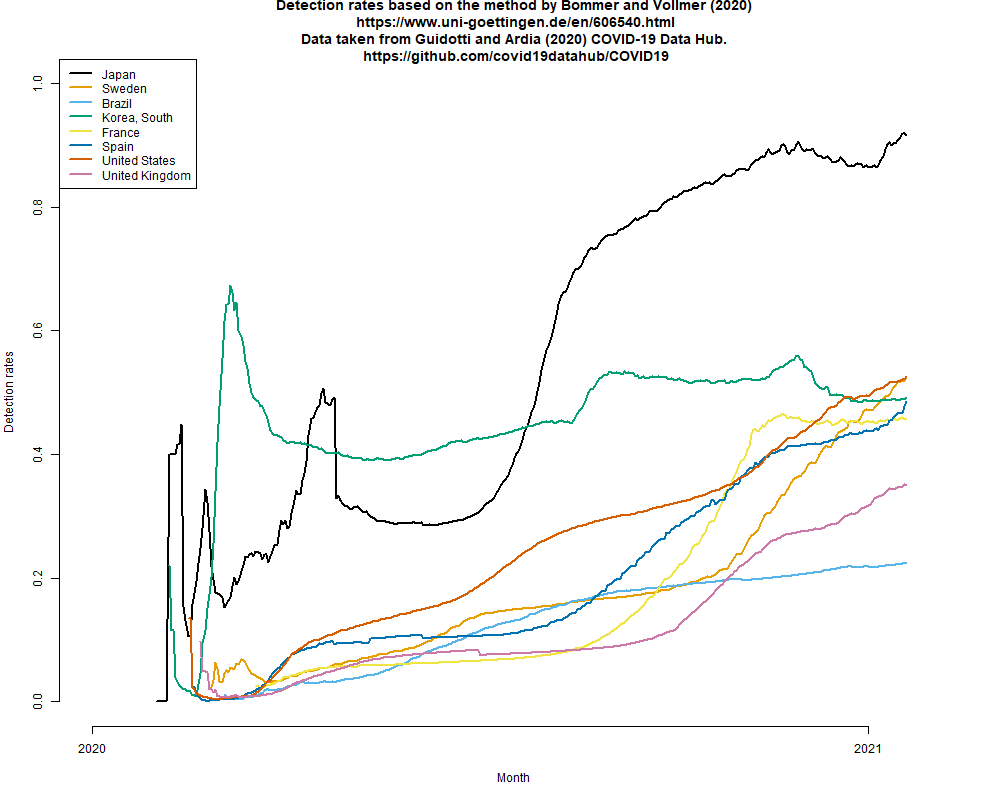
そこでさらに考えてみる。東洋経済サイトによると,年始の数日を除く12月下旬と1月上旬に1を超えていた見かけの(年始の数日に1を切っているのが検査数に依存する見かけの数である証拠)実効再生産数が,現在は0.8くらいになっているが,既に下げ止まっている。接触者追跡を縮小した効果は,過去の検出のうちどれくらいが現在できていない追跡からの検出なのかという割合を計算すればわかるはずなので(そのデータを探すのが最も面倒なところだし,入手可能かどうかわからないし,時間も気力も無いので自分にはできないが),その値で検出数に基づく簡易推定で得られる実効再生産数を割れば,(簡易推定のままではあるが)追跡されずに広がっている部分も含めた実効再生産数が計算できるはず。それが1を超えていたら,新規感染報告数は1月上旬より減っていても,実は新規感染者は増え続けていて,緊急事態宣言をしていてもいつか報告数も増え始めるはず。1を超えていなくても0.95とかだったら,あと1ヶ月緊急事態宣言を続けてもあまり新規感染報告数が減らないことになる。誰かやっている気もするが見つからない。(2021.2.3)
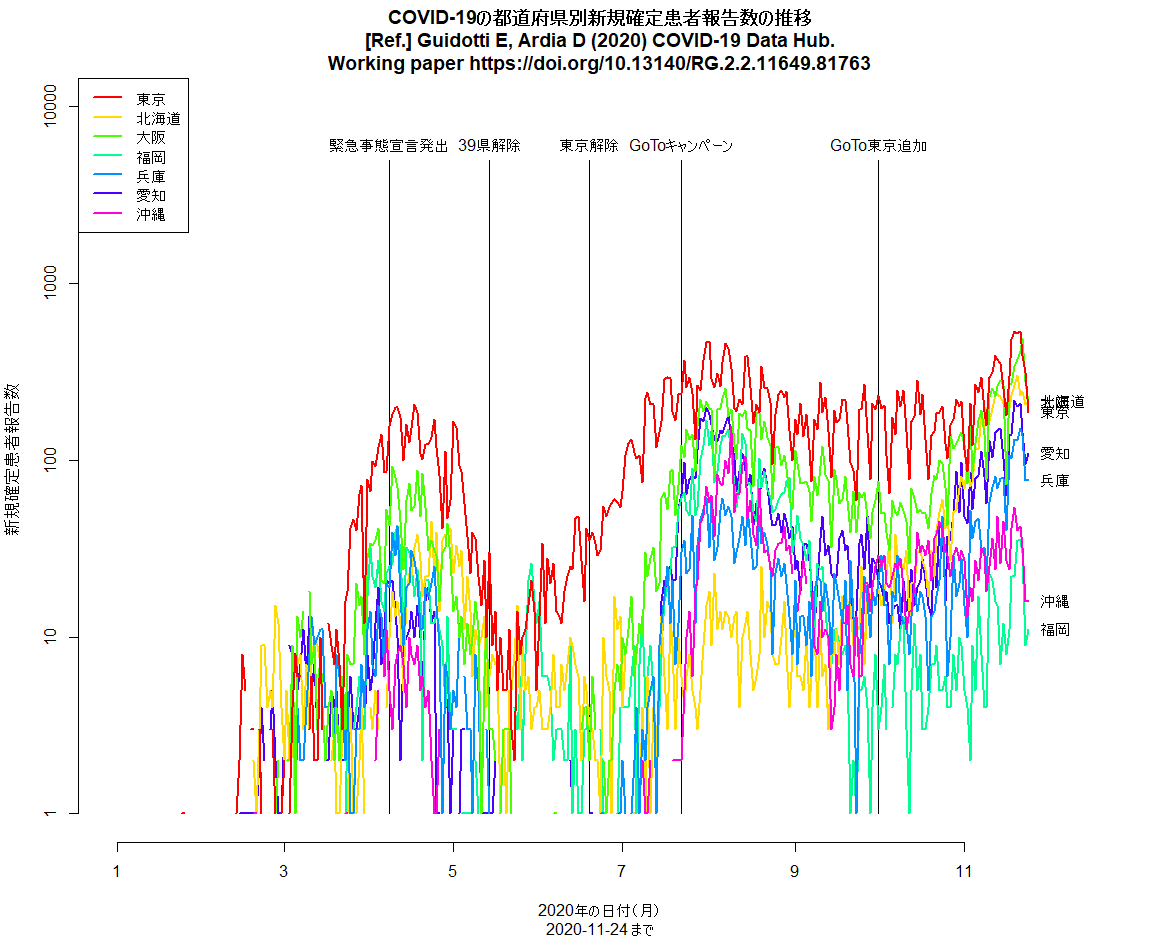
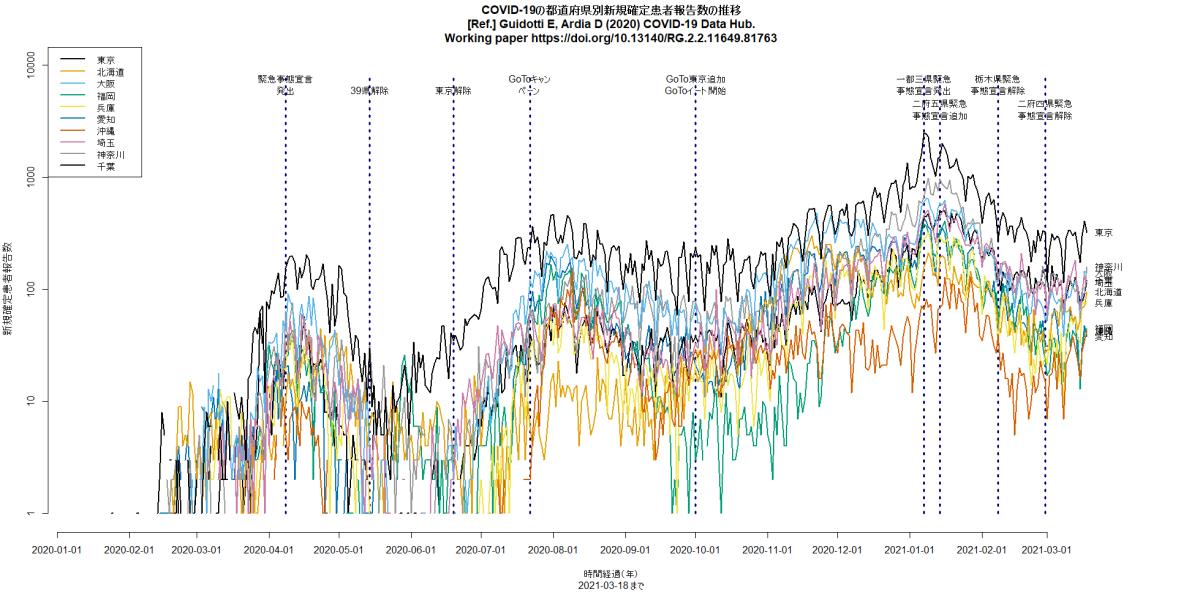
都道府県別新規感染報告数の推移を描くコードに2月末の6府県緊急事態宣言解除の縦線を入れて再実行してみた。3月に入ってから下げ止まりどころか増加傾向なのは(緊急事態宣言継続中の東京都も含めて)明らかなように思うし,おそらく接触者追跡を制限したことによって把握された新規感染者が減ったように見えても,追跡されていない経路で増えた感染者が徐々に把握されるようになってきたとみるのが自然だと思う。(2021.3.19)
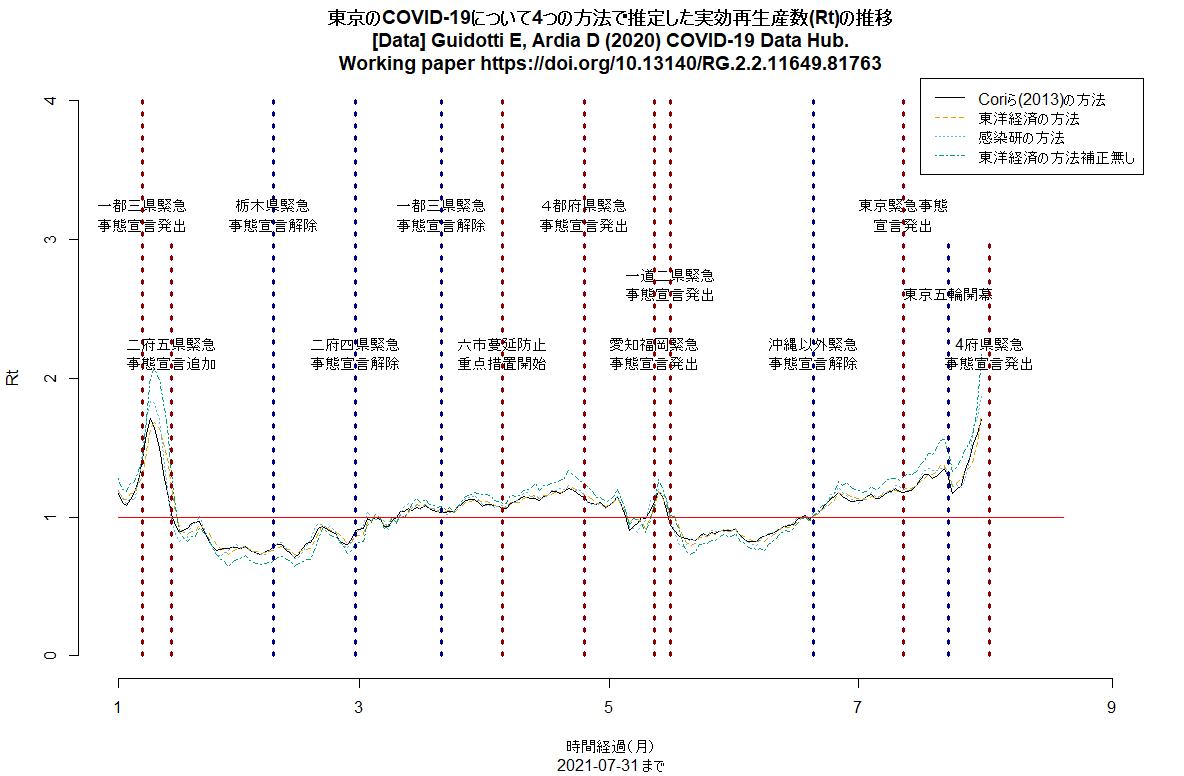
感染研のサイトにCOVID-19感染報告者数に基づく簡易実効再生産数推定方法(2021年6月29日)という記事が載っていた。「直近7日間の新規陽性報告者数/(世代時間)日前7日間の新規陽性報告者数」という,Bonifazi G et al. "A simplified estimate of the effective reproduction number Rt using its relation with the doubling time and application to Italian COVID-19 data"(Eur. Phys. J. Plus,2021年4月11日)が提案した方法で,世代時間を5日として計算すると,Cori A et al. "A New Framework and Software to Estimate Time-Varying Reproduction Numbers During Epidemics"(AJE,2013年9月15日)で提案されている方法で推定されたRtと良く一致するRtが簡単に得られたと書かれている。報告日ベースであるためのバイアスと,すべての感染者で世代時間が同じと仮定している方法であるという制約はあるが,簡単な計算で毎日Rt推定値を更新できるので自治体などの役に立つだろうとのこと。Rtの簡易推定法というと,2020年5月12日に行われた公開セミナーで西浦さんが(直近7日間の新規報告患者数/その前7日間の新規報告患者数)が近似的に使えるかもしれないと言われていたが,その後,東洋経済にアドバイスした方法は,(直近7日間の新規陽性者数/その前の7日間の新規陽性者数)^(平均世代時間/報告間隔);平均世代時間は5日間,報告間隔は7日間と,若干変わっていた。今回感染研のレポートに出ている方法は,そのどちらとも微妙に違っている(そんなに大きくは違わないようだが)。ちなみに,西浦さんが厚労省に提示している資料や論文では,ここに書かれているように,世代時間は4.8±2.3日,潜伏期間は5.6±3.9日とし,右側打ち切りを考慮し,Rのsurveillanceパッケージを使って確定診断日から発症日を逆推定した上で発症日から感染日を逆推定し,感染日から二次感染日までの間隔である世代時間について再生方程式を立ててRtを計算しているので,報告遅れも世代時間のばらつきも考慮できているが,コンピュータなしでは計算できないし,何日も前の値までしか推定できないという制約がある。以上のアルゴリズムをRのコードにして(若干ちゃんと詰めずに書いている部分があるので,間違いを含んでいる可能性があり,あまり信じないで欲しいが)東京のデータについて計算させてみたところ,どの方法の結果もまあまあ合っていて,6月に緊急事態宣言を解除した頃から,どの方法でもRtは1を超えている。(2021.7.2)
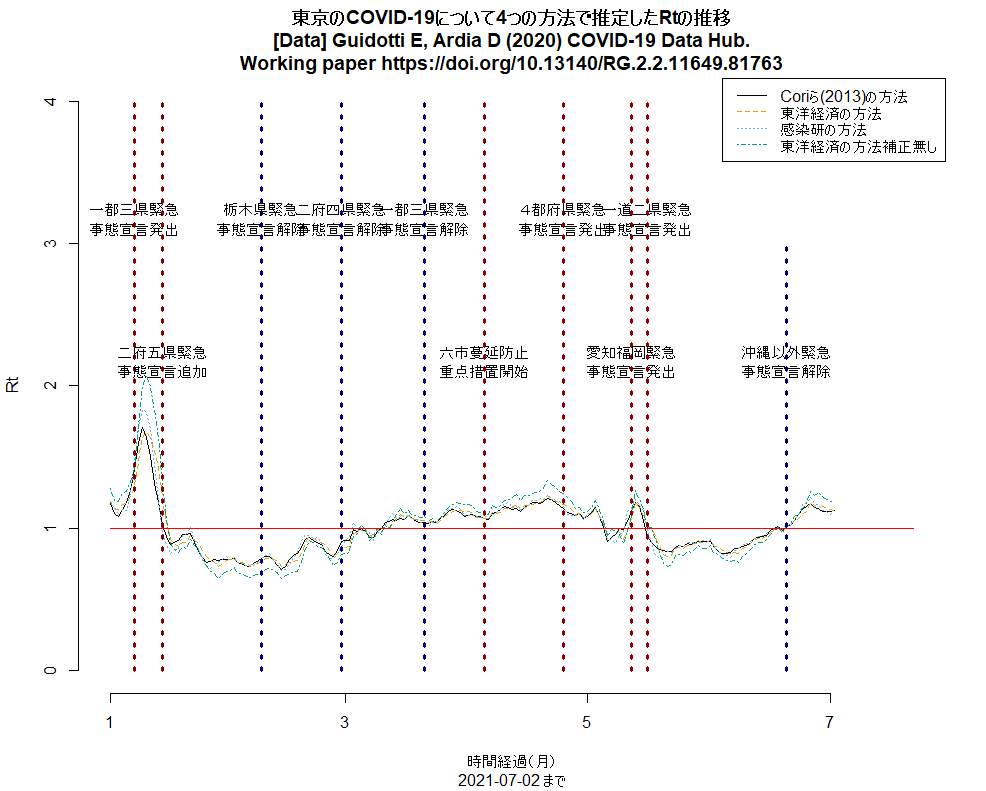
明日からの東京緊急事態宣言の線を入れるコードを追加して(東京のRt推移,日本の都道府県別新規感染者数推移),実行させた結果をtweetした。東京のRtは明らかに上昇中(下図参照)。(2021.7.11)
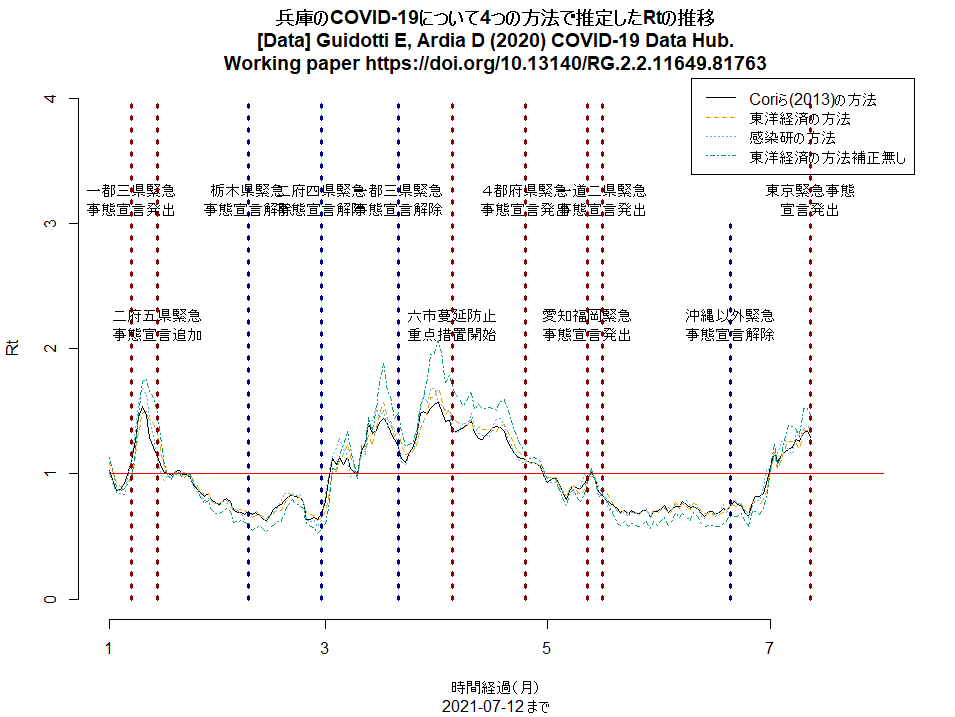
神戸市を含む兵庫県の5区域で昨日から「まん延防止」が解除され,「感染リバウンド防止」という,より緩い制限に移ったが,この文書に「下げ止まり」とあるのは認識が甘く,緊急事態宣言解除以降,大阪同様にRtは上昇中で,既に1を超えている(このRコードで描ける下図参照。このコードは都道府県名を変えるだけでどの都道府県にも使えるはず)。このまま緩めていたらすぐに再び3桁/日になりそう。(2021.7.13)
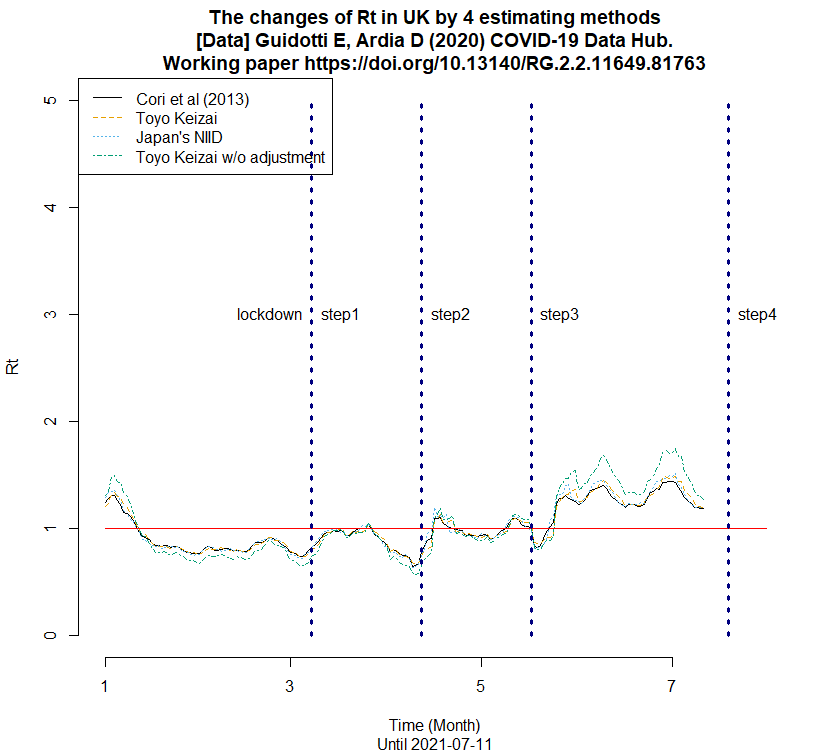
UKのRtの推移を描くRコードも作って走らせてみた(下図)。(2021.7.13)
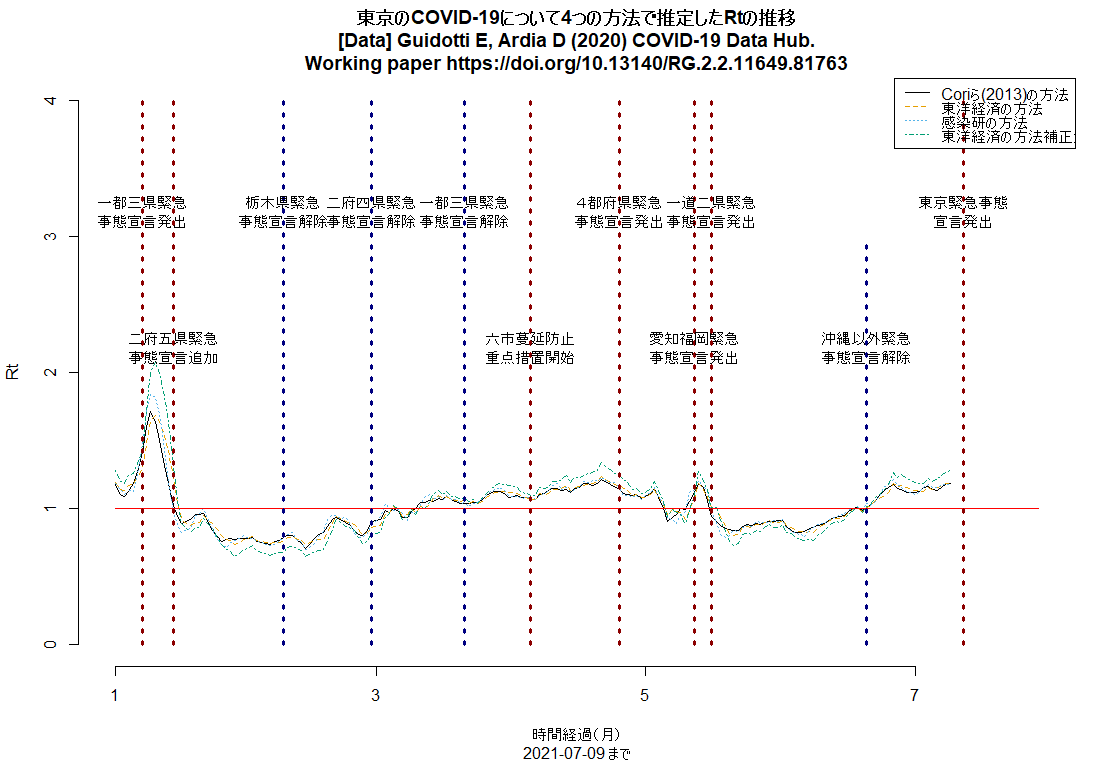
東京のRt推移について,若干Rコードをアップデートして再び走らせてみた。(1)第3波と第4波では緊急事態宣言で(感染から報告までのラグを考えるとそのやや前から)Rt低下,(2)第5波への緊急事態宣言は無効,(3)五輪開幕から(たぶんその少し前からの開放感と期待感とデルタ株で)Rt急上昇中,が見える。(2021.8.1)
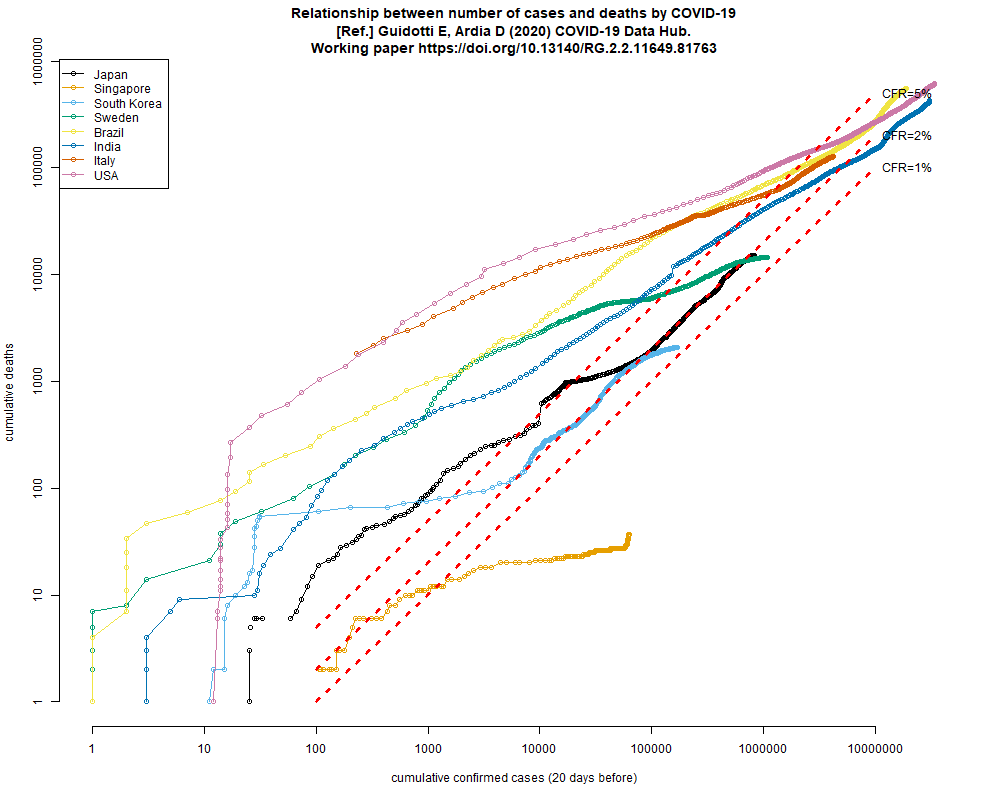
以前も示したことがあるが,このコードで累積死亡数と20日前までの累積確定感染者報告数の関係を両対数グラフにプロットすると,ごく大雑把なCFRを見積もれる(下図)。シンガポールを除けばどの国でもだいたい1%から5%くらいの幅に収束しそうで,日本はこのところ2%くらい。まだ死者が急増していなくても甘く見てはいけない。(2021.8.2)
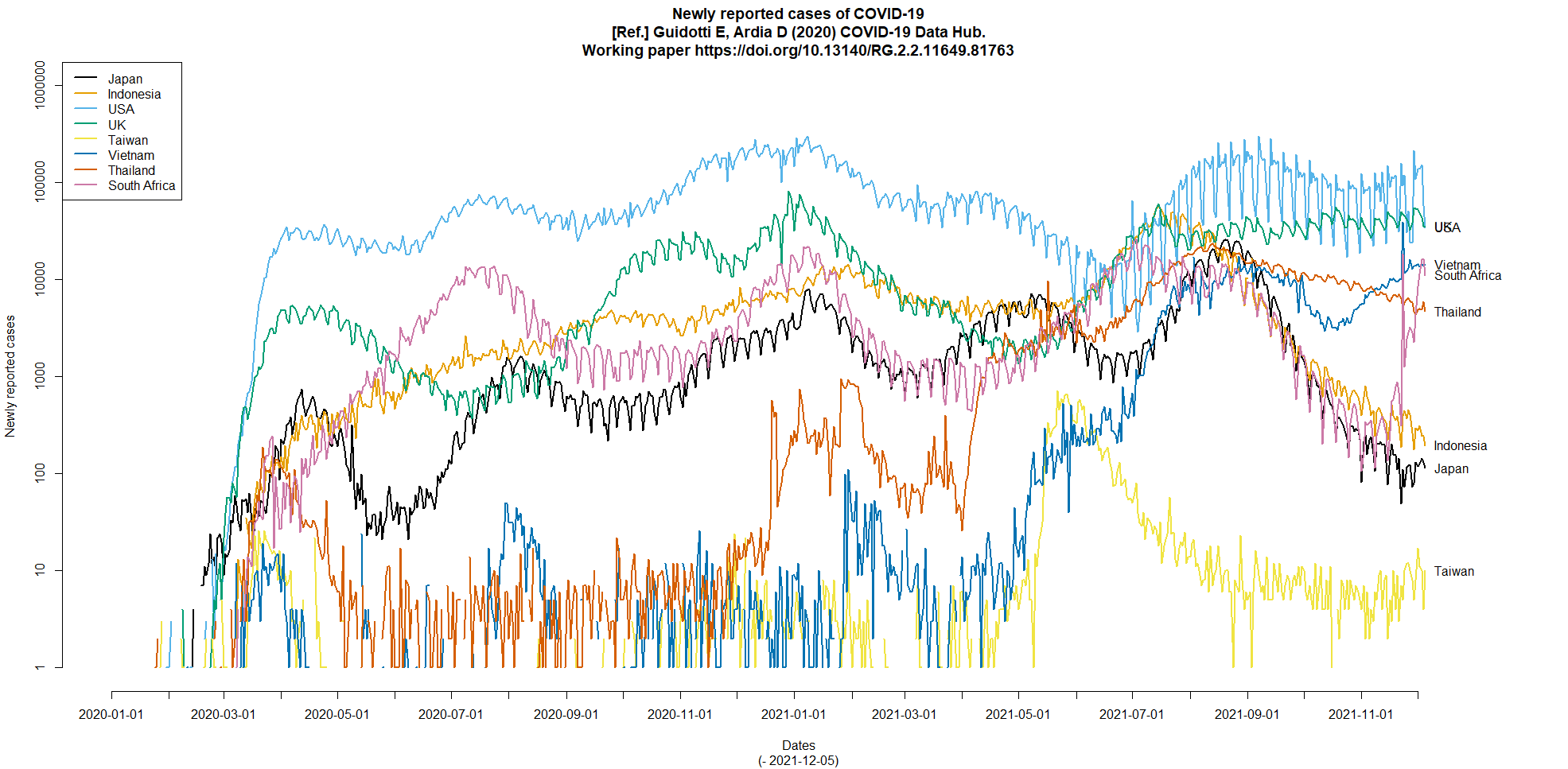
COVID19パッケージのアルファベット3文字国名コードを示す変数名がidからiso_alpha_3という変数名に変わっていたので(変数名idはまだ存在しているが,意味不明の文字列になっていた),かつて作ったCOVID-19の毎日の感染確定報告数の推移を示す折れ線グラフを描くコードが動作しなくなっていた。変数名を書き換え,南アフリカを追加したコードに書き換えてグラフを作ってみた。8月下旬から11月上旬くらいまで日本とインドネシアと南アフリカがほぼ同じような動きなのに,11月中旬以降の南アフリカの報告数急増が凄い。B.1.1.529変異株が南アフリカからWHOに報告されたのは11月24日だったが,それ以前から増えていたということだろう。(2021.12.8)
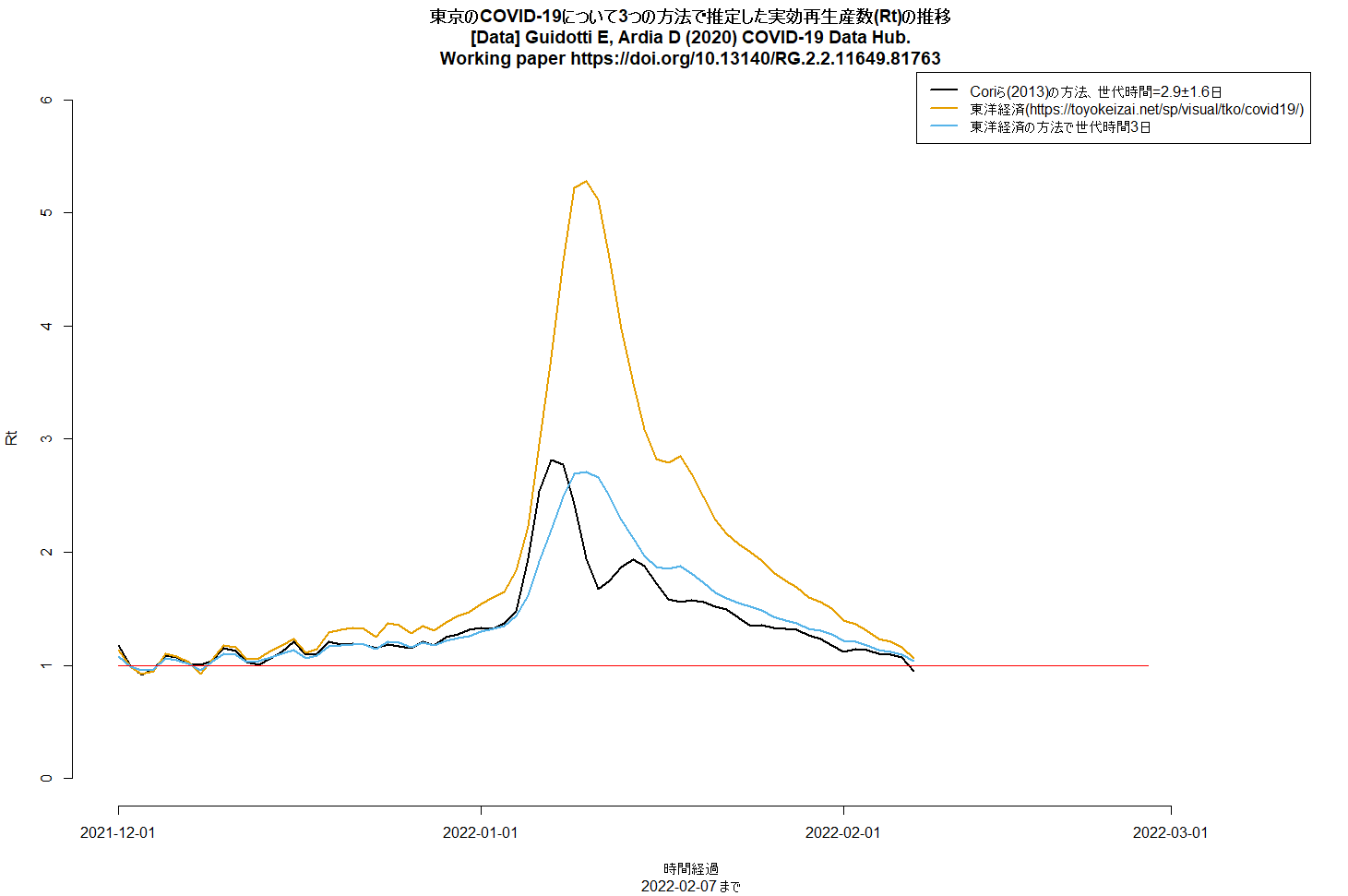
東洋経済のサイトのRtの計算が、現在でも平均世代時間5日で計算されているため、ピークが異常に高くなってしまっている。オミクロン株が主だとすると、例えば韓国の接触追跡データから平均世代時間を推定したSong JS, Lee J, Kim M, Jeong HS, Kim MS, Kim SG, et al. Serial intervals and household transmission of SARS-CoV-2 Omicron variant, South Korea, 2021. Emerg Infect Dis. 2022 Apr [2022年2月7日参照].(Emerging Infectious DiseasesのResearch Letter、2022年2月2日)によると、オミクロン株の平均発症間隔が2.9±1.6日、中央値が3日なので、これが世代時間の代わりに使えるとしたらCoriら(2013)の方法でRtが推定できるし、東洋経済の方法で平均世代時間3日にして計算することもできる。やってみると、ピークのRtが3より少し低いくらいの値になる。また、Coriらの方法で最後Rtが1を下回っているように見えるのは検査の遅れによる過少報告の影響の可能性が高いので、収束し始めたと考えるのは早計だろう。
Fox SJ et al. "Real-time pandemic surveillance using hospital admissions and mobility data"(ProNAS、2022年2月15日号)は、SEIRのバリエーション(発症前感染力あり状態とか、無症状感染力あり状態といったコンパートメントを考えている)のモデルを開発し、テキサス州オースティンでの入院データと携帯電話の移動データを使って、COVID-19のリアルタイム伝播と入院予測システムを作ったという報告。Appendixにモデルの詳細が書かれていて、データとコードはGitHubで公開されている。3/4はRで、1/4はC++で開発されたもののようなので、後で暇があったら試してみよう。
新規感染者数と死者数の推移は、やはり20日くらいずれているようなので、累積死者数と20日前までの累積感染者数の両対数グラフからラフなCFRの推移をみるというやり方は悪くないアイディアだったと思う。
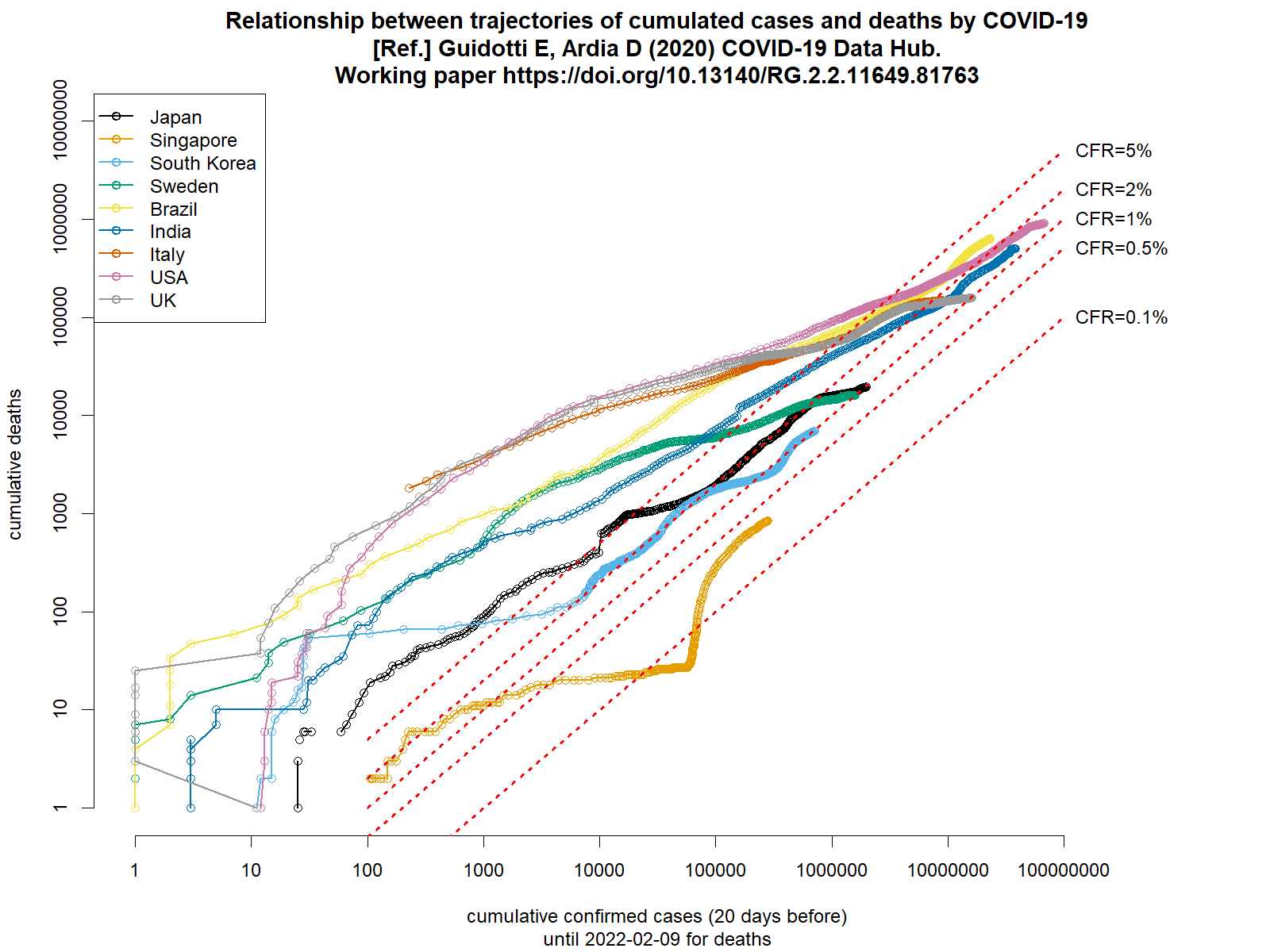
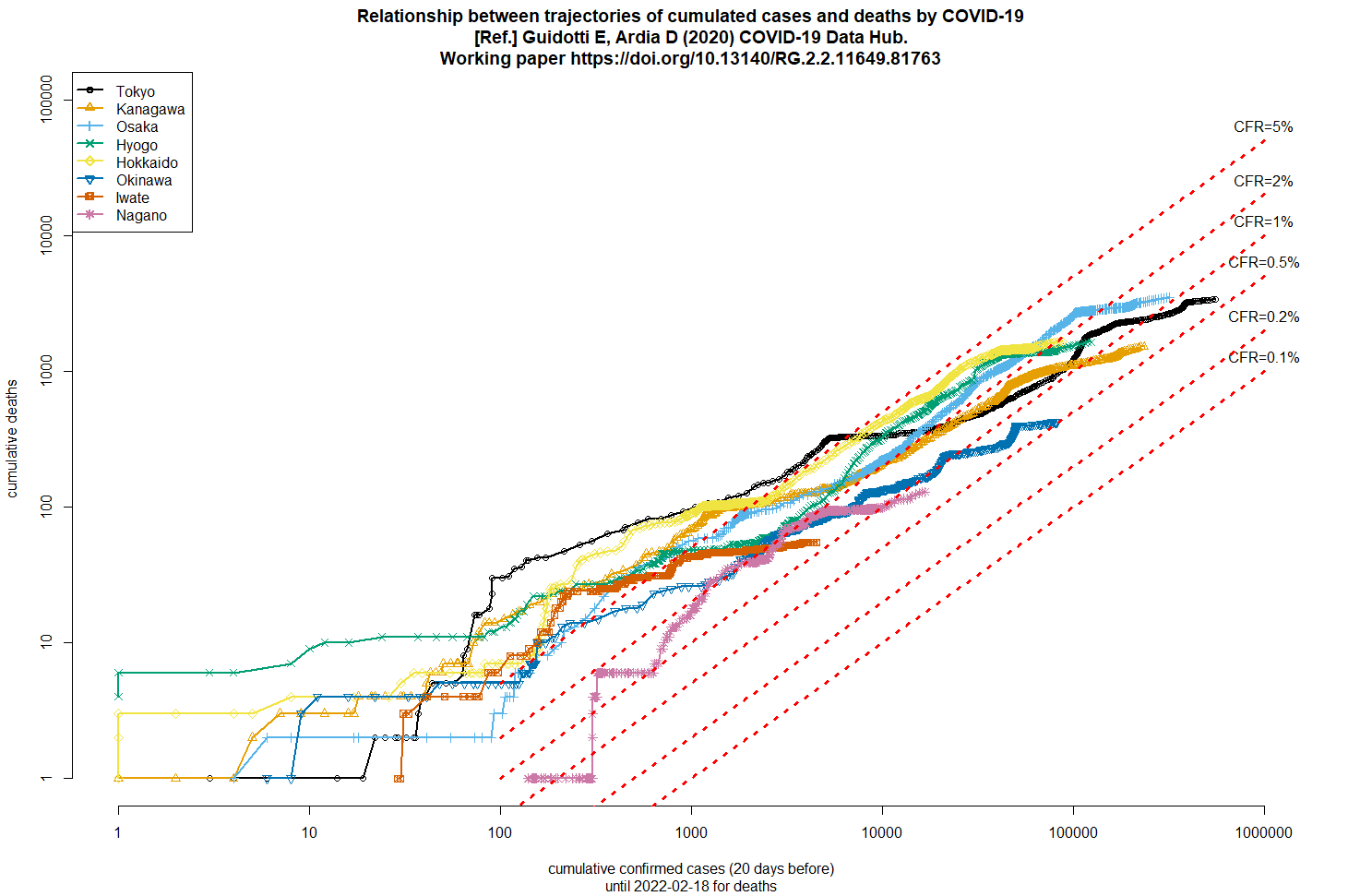
2022年2月16日の早朝にtweetした図を作るRコードとグラフを、ここにも貼っておく。tweetしたときの傾向は今も続いていて、大阪や兵庫の簡易推定CFRは1%を上回っているが、沖縄は0.5%を切った。感染者が少なくて簡易推定CFRが1%を上回っている岩手は、たぶん高齢の感染者が多いのではないか。
R-3.6.3リリース(2020年2月6日)
R-3.6.3が2月29日(R-1.0.0リリースから5回目の=閏年にしかない日なので20周年だが=記念日)にリリース予定とのこと。ニックネームは"Holding the Windsock"。
既に予告されていた通り,Rの5回目(20年目)の誕生日である今日,R-3.6.3 "Holding the Windsock"のソースバージョンがリリースされた。
Correspondence to: minatonakazawa[at]gmail.com.