【第388回】 もう半年経過(2013年7月1日)
- 6:00に「Awakening」で起床。もう今年も半分過ぎてしまった。入浴してから玄米ご飯に豚肉野菜カレーを食べ,講義準備の続き。2番のバスで出勤予定……だったが,講義準備を途中で止められなかったので3番のバス。
- 10:00からアリムさんの論文指導,13:00からと18:30から他領域の大学院生の統計解析相談,14:50から講義と今日も忙しかった。講義室のプロジェクタのランプが切れていて映写できず,ポータブルのものを教務係に借りに行って15分近く損したので,今日も講義には余裕がなかった。
- 講義資料を準備する過程で,様々な物質のLD50を示そうと思い,化学物質の分子模型を描画できるソフトを探してみた。昔はISIS Draw(だったか)を使って部品を組み合わせて作ったものだが,今では大抵の低分子は日本化学物質辞書Webから*.molという形式で,タンパクなら日本蛋白質構造データベース(pdbj)から*.entという拡張子のついた形式でダウンロードできる。
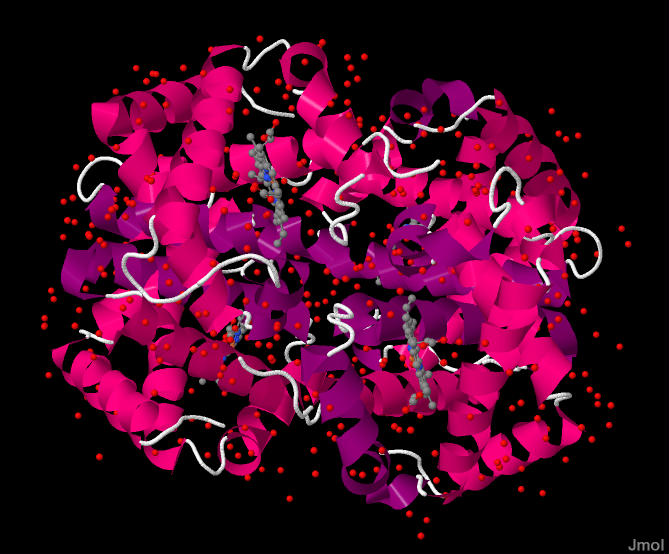
- 例えば後者からヒトヘモグロビンを検索して,デオキシヘモグロビンであるpdb1a3n.entをダウンロードし,それをJmolで開くだけで上の図ができてしまうのだ。Jmolは元素を付け加えたり削除したり種類を変えたりもできるし,マウスで回転もできるし,図の見せ方も「棒球」を選ぶと下の図のようにもできる。保存形式もいろいろあるのだが,とりあえずjpgで保存してみた。

- fmsbライブラリのclifetable()やlifetable()の内部ではcumsum()関数を使っているため,NAが入っているとTxとexが計算できない。とりあえずの対処としては,NAでないところだけ使えばいい。例えば,library(fmsb)に続けて,clifetable(Jlife$qx2010M[!is.na(Jlife$qx2010M)])とすれば計算できる。ここでa0=0.1, a1〜a4=0.4として計算したければ,lifetable(qxtomx(Jlife$qx2010M[!is.na(Jlife$qx2010M)]), mode=2, class=1)とすればいい。若干結果が変わるが,これはaxを使ったからである。ちなみにPreston SH et al.(2001)のテキストに載っている男性のaxを使うには,lifetable(qxtomx(Jlife$qx2010M[!is.na(Jlife$qx2010M)]), mode=6, class=1)とすればいい……と思ったら,mode=6とmode=7がバグっていた。直さなくては。
- 直通終バスで帰り,昨夜作ったカレーを食べ尽くした。
△Read/Write COMMENTS
▼前【387】(結局今日も講義準備(2013年6月30日) ) ▲次【389】(今日も3番のバス(2013年7月2日) ) ●Top